关键词:机器学习,蛋白质结构预测,聚类算法,自然演化,蛋白质宇宙,机器学习,生物信息学
Nature 速递:在已知蛋白质宇宙尺度上聚类预测的结构
论文标题:Clustering-predicted structures at the scale of the known protein universe
斑图链接:https://pattern.swarma.org/paper/566b6b70-5289-11ee-8c54-0242ac17000d
原文链接:https://www.nature.com/articles/s41586-023-06510-w
蛋白质在所有细胞过程中都起着关键作用,其结构对于理解其功能和演化至关重要。基于序列的蛋白质结构预测的准确性不断提高,AlphaFold 数据库中提供了超过2.14亿个预测结构,而以这种规模研究蛋白质结构需要高效的方法。
文章开发了一种基于结构比对的聚类算法——Foldseek cluster,可以对数亿个结构进行聚类。使用这种方法,作者对 AlphaFold 数据库中的所有结构进行了聚类,识别出230万个非单体结构聚类,其中31%没有注释,代表可能是以前未描述的结构。没有注释的聚类只具有少量代表,仅覆盖 AlphaFold 数据库中所有蛋白质的4%。演化分析表明,大多数聚类具有古老的起源,但4%似乎是物种特定的,代表预测质量较低或新生基因的示例。
文章还展示了如何利用结构比较来预测结构域家族及其关系,识别远程结构相似性的示例。
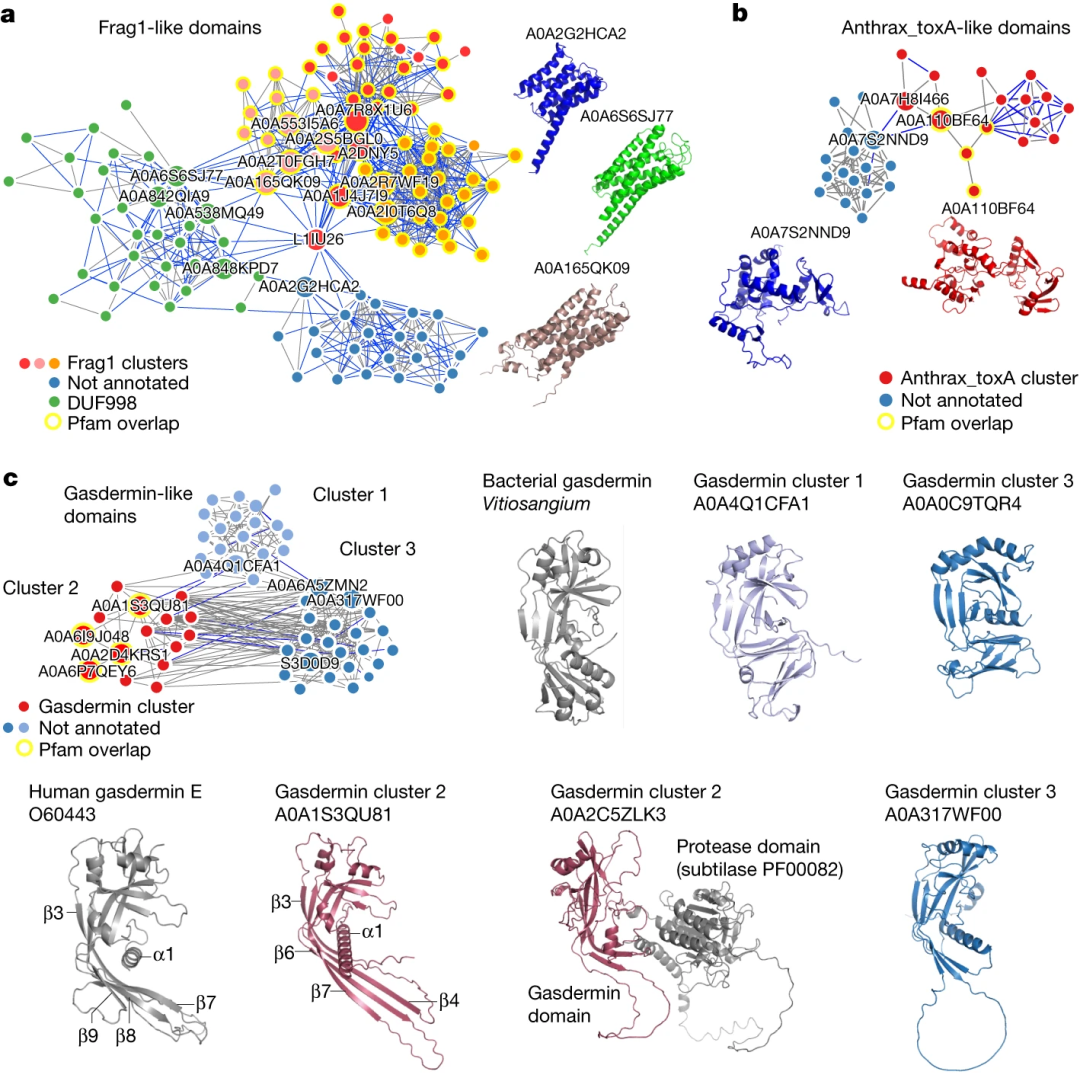
图2 与已注释的域家族存在结构相似性的未注释域家族示例
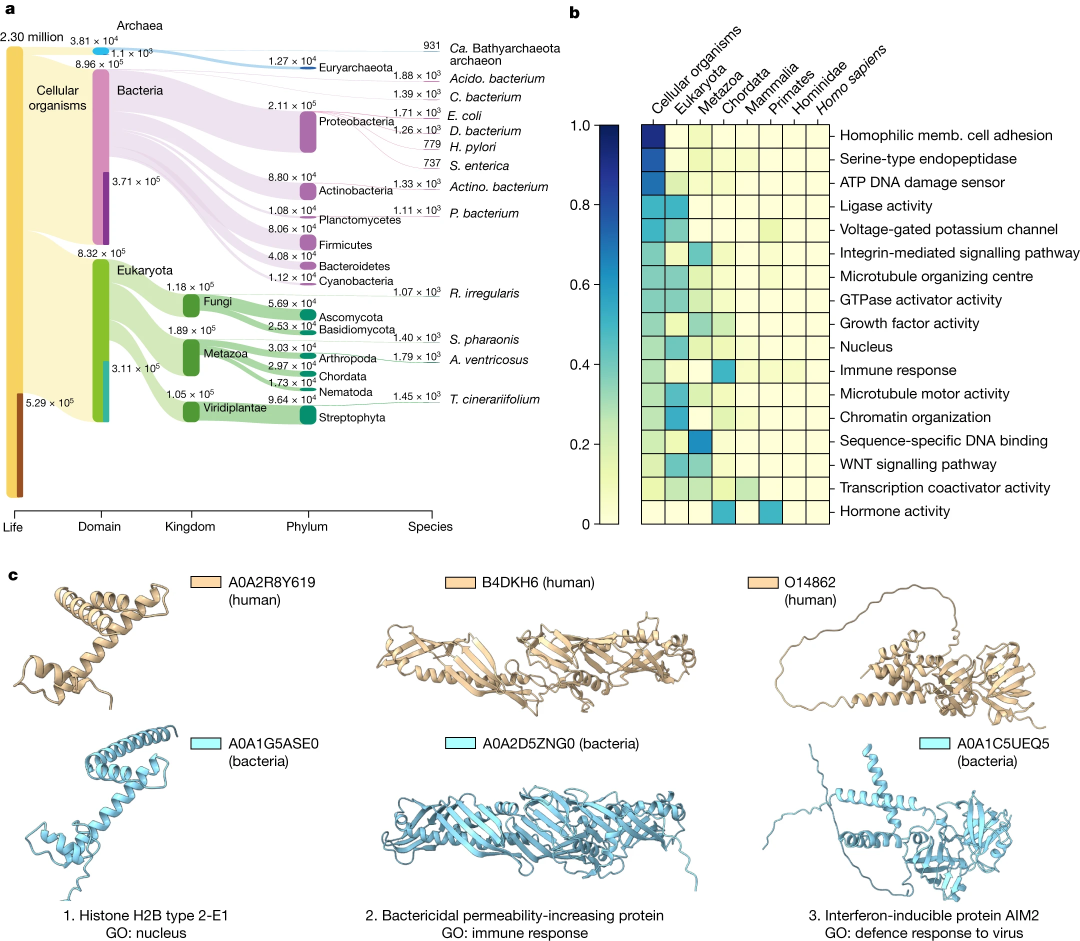
基于这些分析,作者确定了一些人类免疫相关蛋白质的潜在远程同源性在原核生物中,这说明这一资源在跨生命树研究蛋白质功能和演化方面的价值。
Nature速递:揭示自然蛋白质宇宙中的新家族和新折叠
论文题目:Uncovering new families and folds in the natural protein universe
斑图地址:https://pattern.swarma.org/paper/4b9269c4-5289-11ee-8c54-0242ac17000d
论文地址:https://www.nature.com/articles/s41586-023-06622-3
我们正进入蛋白质序列和结构注释的新时代,通过 AlphaFold 数据库可以获得数亿个预测的蛋白质结构。这些模型几乎覆盖所有已知蛋白质,包括那些使用标准的基于同源性的方法难以注释其功能或假定的生物学作用的蛋白质。
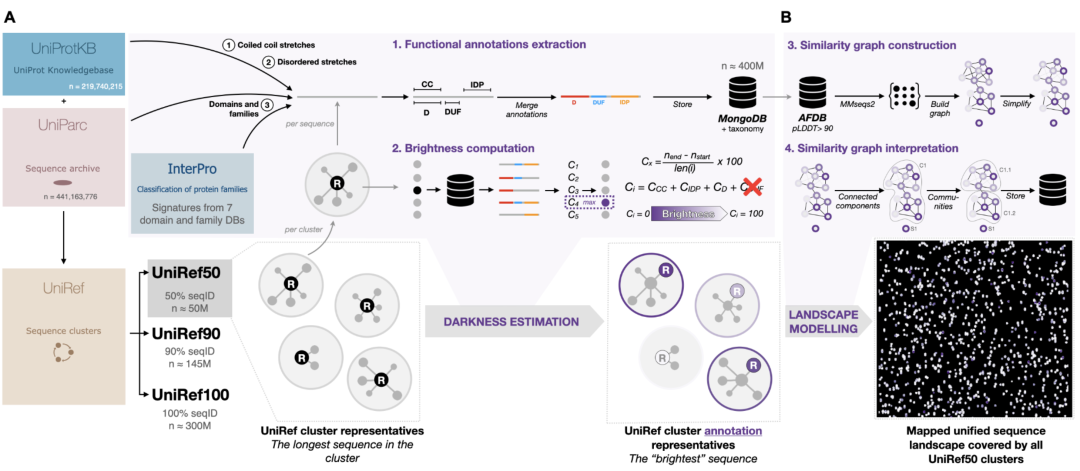
图1. UniProt 和 AlphaFold 数据库中集合、分类和映射未知功能蛋白质的工作流。
这项工作研究了 AlphaFold 数据库在多大程度上以高预测精度照亮了自然蛋白质宇宙的“暗物质”。研究进一步描述了这些模型覆盖的蛋白质多样性,以带注释的互动序列相似性网络的形式呈现,可访问https://uniprot3d.org/atlas/AFDB90v4 。
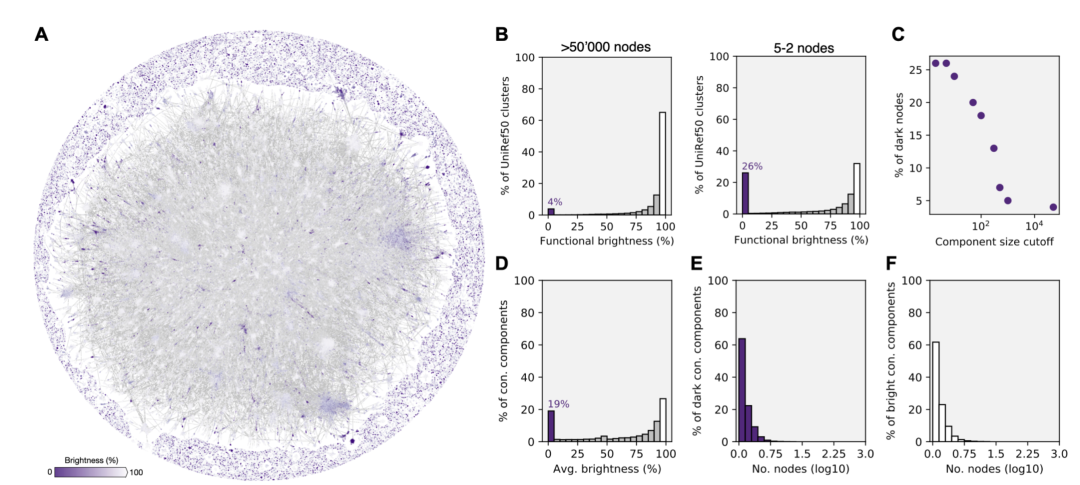
图2. AFDB(AFDB90)中超过600万个 UniRef50 簇代表蛋白质的大规模序列相似性网络,具有高度预测准确性的模型。
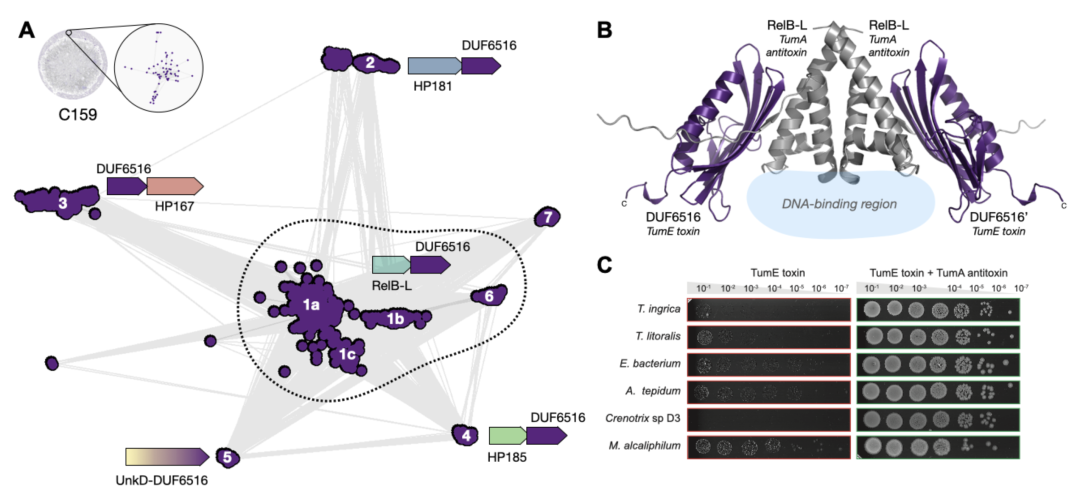
通过从序列、结构和语义角度搜索新颖性,研究揭示了β-花瓣折叠结构,向 Pfam 数据库中添加了多个蛋白质家族,并在实验中证明其中之一属于新的翻译靶向毒素-抗毒素系统超家族 TumE-TumA。
图3. 关联部分159是毒素-抗毒素超家族 TumE-TumA 中的新毒素。
这项工作强调了在识别、注释和优先考虑新蛋白质家族方面大规模努力的价值。利用蛋白质生物信息学领域的最新深度学习革命,我们现在能过以前所未有的规模照亮蛋白质宇宙的未知领域,为生命科学和生物技术创新铺平道路。
大模型与生物医学:
AI + Science第二季读书会启动
详情请见:
大模型与生物医学:AI + Science第二季读书会启动
推荐阅读