Nat. Commun. 速递:基于异质图 transformer 的单细胞生物网络推理

关键词:生物网络,基因调控网络,图神经网络,机器学习


论文题目:Single-cell biological network inference using a heterogeneous graph transformer 论文来源:Nature Communications
论文链接:https://www.nature.com/articles/s41467-023-36559-0
单细胞多组学(Single-cell multi-omics, scMulti-omics)允许同时对多种组学数据进行定量分析,以捕捉复杂的分子机制和细胞异质性的复杂性。现有的工具不能有效地推断不同细胞类型的活性生物网络以及这些网络对外部刺激的反应。
Nature Communications 的这项研究提出了利用单细胞多组学数据进行生物网络推理的算法架构 DeepMAPS。它在一个异构图中对单细胞多组学数据建模,并使用一个 multi-head graph transformer(HGT)在局部和全局上下文中以一种鲁棒的方式学习细胞和基因之间的关系。基准测试结果表明,DeepMAPS 在细胞聚类和生物网络构建方面的表现优于现有工具。它还展示了在肺肿瘤白细胞 CITE-seq 数据和匹配的弥漫性小淋巴细胞淋巴瘤的单细胞 RNA 和单细胞 ATAC 数据中推导细胞类型特异性生物网络上,表现优于其他工具。此外,研究还部署了一个可提供多种功能和可视化的 DeepMAPS 网络服务,以提高单细胞多组学数据分析的可用性和可重复性。
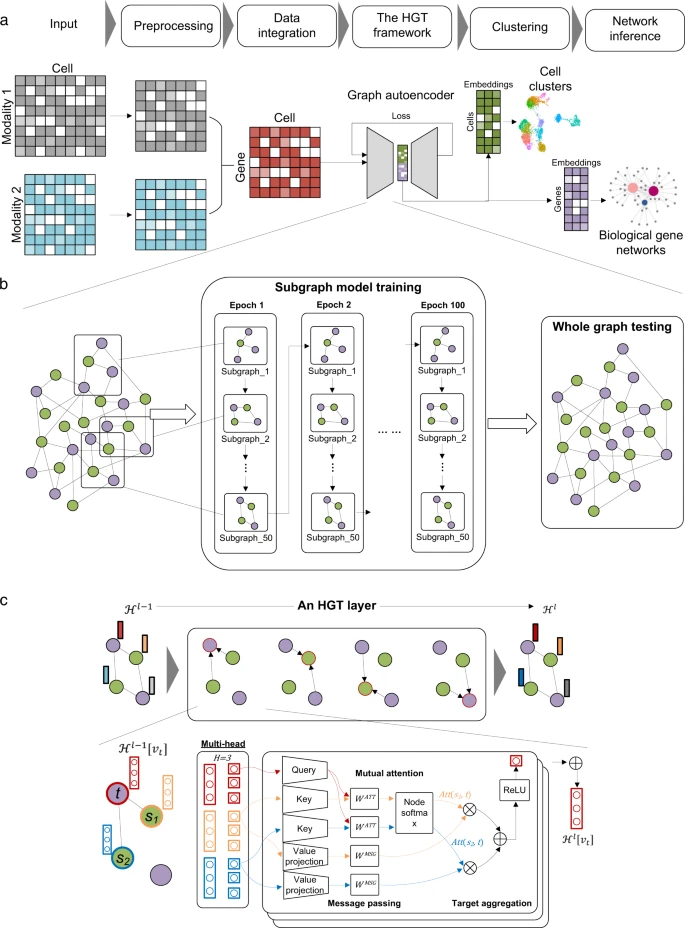
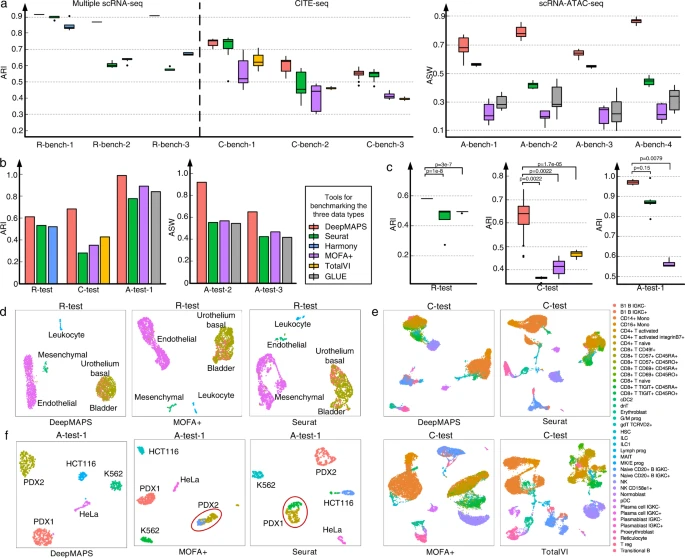
图2. DeepMAPS 在细胞聚类上的表现评价
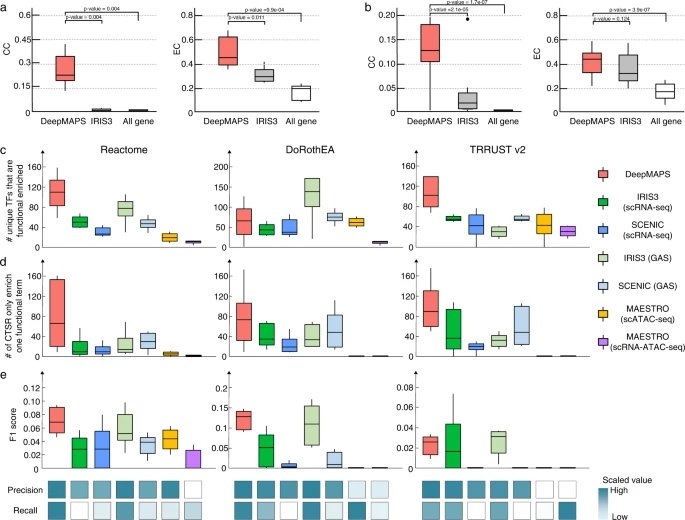
图3. 在基因关联网络推断任务上,DeepMAPS的性能评价及和其他方法的对比
图神经网络读书会
推荐阅读










