跨越5亿年的生命设计革命:Science发布大语言模型生成全新荧光蛋白


论文题目:Simulating 500 million years of evolution with a language model 发表时间:2025年1月16日 论文地址:https://www.science.org/doi/10.1126/science.ads0018 期刊名称:Science
研究团队将蛋白质的序列、结构和功能编码为离散标记(Token),构建了跨模态的Transformer架构:
1. 结构化难题攻克:将三维原子坐标压缩为离散标记(Structural token),通过几何注意力机制实现空间推理,突破传统扩散模型的复杂度限制(结构重建误差<0.5Å)。
2. 全局推理训练:采用遮蔽语言模型(Masked language modeling),随机遮蔽任意模态数据,迫使模型通过其他信息补全,例如通过功能关键词反推结构。
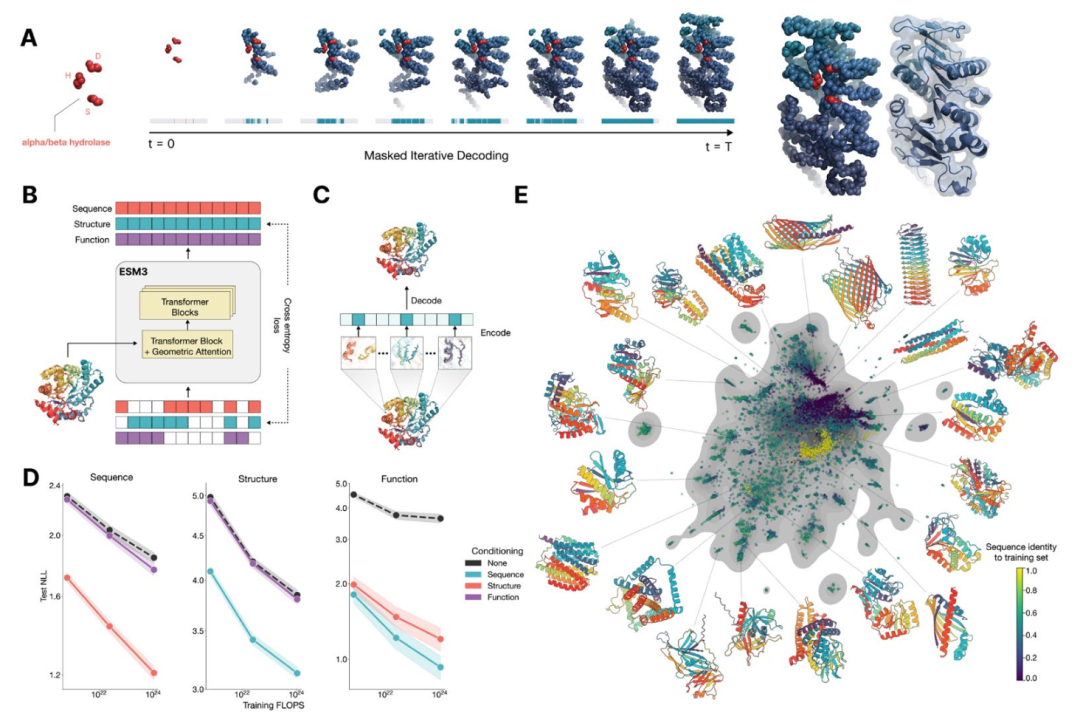
图 1. ESM3 是一种生成式语言模型,能够对蛋白质的序列、结构和功能进行推理。(A)ESM3 的迭代采样。生成一种α/β水解酶。序列、结构和功能都可以用作提示模型。在每个时间步 t,对部分掩码位置进行采样,直到所有位置都未被掩码。(B)ESM3 架构。序列、结构和功能在输入和输出端均表示为离散标记的轨道。该模型由一系列的 Transformer 块组成,所有轨道都在一个单一的潜在空间中融合;第一个块中的几何注意力允许基于原子坐标进行条件设置。ESM3 通过预测掩码标记进行监督训练。(C)结构标记化。每个氨基酸周围的局部原子结构被编码为标记。(D)模型在三个规模上进行训练:14 亿、70 亿和 980 亿参数。测试集上负对数似然(平均于掩码率)作为训练 FLOPs 的函数显示了对每个输入轨道进行条件设置的响应,随着 FLOPs 的增加而提高(95%置信区间)。(E)ESM3 980 亿参数模型的无条件生成(按颜色区分)与训练集中最接近序列的序列一致性,通过 ESM3 嵌入,并通过 UMAP 投影,同时还有从 UniProt 中随机抽取的序列(以灰色显示)。生成的序列多样、质量高,并涵盖了天然序列的分布。
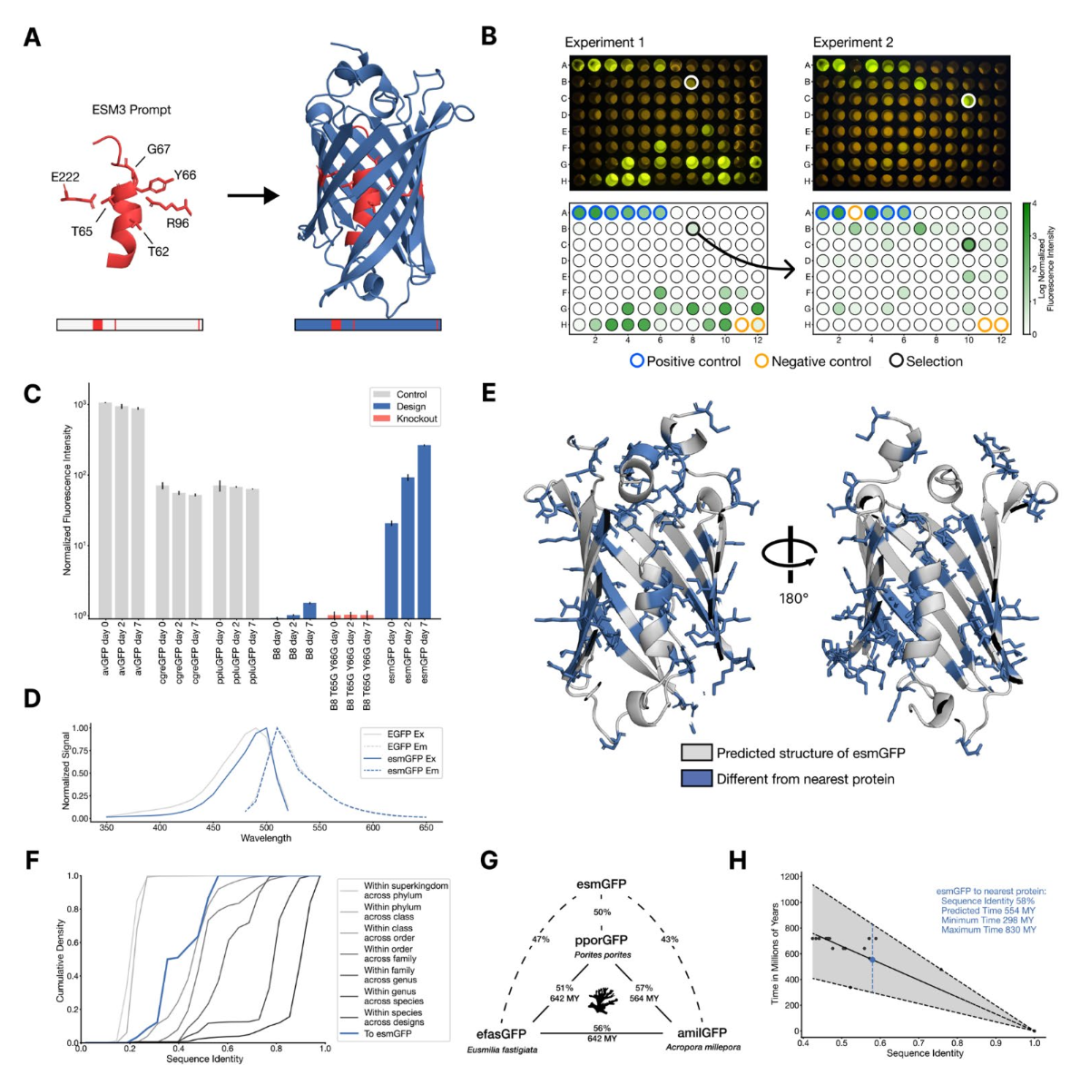
图 2. 通过一系列的思考生成一种远距离荧光蛋白。(A)我们用形成和催化发色团反应所需的残基序列和结构以及天然绿色荧光蛋白(GFP)部分中央α螺旋的结构来提示 ESM3(左)。通过一系列的思考,ESM3 生成设计候选(右)。(B)在两次实验中,ESM3 找到了一种远离其他已知 GFP 的明亮 GFP。我们在大肠杆菌裂解液中测量了荧光。上排,平板照片。下排,平板读取器荧光定量。已知 GFP 的阳性对照用紫色圆圈标记,没有 GFP 序列或没有大肠杆菌的阴性对照用红色圆圈标记。在第一次实验(左)中,我们表达了具有不同序列同源性的设计。在 B8 孔(底部黑色圆圈,顶部白色圆圈突出显示)中出现了一个值得注意的设计,其与已知荧光蛋白的序列同源性较低(57%)。我们从 B8 孔中的蛋白质继续进行第二次实验(右)。在 C10 孔(与已知荧光蛋白的序列同源性为 58%,黑色圆圈)中出现了一个明亮的设计。(B)我们将其命名为 esmGFP 的是底部为白色圆圈顶部为绿色荧光蛋白的结构。(C)esmGFP 的荧光强度与常见的 GFP 相似。实验 2 中部分蛋白质的归一化荧光强度如图所示。(D)esmGFP 的激发和发射光谱与 EGFP 的光谱重叠。(E)esmGFP 预测结构中中央α螺旋和β桶内部的两个截面图。esmGFP 相对于其最近的邻居 tagRFP 的 96 个突变以蓝色显示。(F)不同分类群中荧光蛋白之间的序列同源性累积密度。esmGFP 与其他所有 FP 的相似程度通常在比较不同目但同属的序列时才会出现。(G)三种典型珊瑚纲 GFP 和 esmGFP 的进化距离(以百万年为单位)和序列同源性。(H)根据 GFP 序列同源性估算的进化距离(以百万年为单位)。我们估计 esmGFP 与已知最近的蛋白质在自然进化中相隔超过 5 亿年。
彭晨 | 编译
大模型与生物医学:
AI + Science第二季读书会
详情请见:
6. 加入集智,一起复杂!










