Barabási组最新:网络科学大幅缩小新冠特效药搜索范围至81种
疫情期间,著名网络科学家 Albert-László Barabási 带领科研团队,运用网络医学的方法预测出能够治疗新冠肺炎的候选药物。该方法构建了蛋白质相互作用网络,从网络结构的层面研究新冠肺炎的特性,根据特性预测候选药物。科研团队目前已购买候选药物在人体组织细胞中进行测试,对效果好的化合物将进行临床测试。近日在 arXiv.org上线的一篇预印本论文中,揭示了该研究成果。

论文题目:
Network Medicine Framework for Identifying Drug Repurposing Opportunities for COVID-19
论文地址:
https://arxiv.org/abs/2004.07229
概述
新冠疫情肆虐全球,形势严峻,人们迫切需要特效药和疫苗来对抗病毒。而传统的药物研发流程周期过长,无法适应对新冠治疗的迫切需求。因此,著名网络科学家Albert-László Barabási 等人认为当下更可行的方式是探索已获美国 FDA(食品药品监督管理局)批准的药物是否具有治疗新冠的功效。经过近 2 个月的努力,网络科学家们成功打通了使用网络医学 (Network Medicine)方法预测治疗新冠候选药物的研究路线。
关于 AL Barabási 的集智百科:
https://wiki.swarma.org/index.php?title=艾伯特-拉斯洛·巴拉巴西_Albert-László_Barabási
蛋白质相互作用网络常用于网络医学研究。该网络中,节点为蛋白质,边为蛋白质间的相互作用关系,如物理结合。人体蛋白质相互作用网络(Human Interactome)揭示了人体内所有蛋白质间的密切联系,是了解人体内生物过程的一张“地图”,也是该论文的基础。人体蛋白质相互作用网络会受到两个外部因素的影响:1. 新冠病毒对于人体蛋白质的影响,2. 药物对于人体蛋白质的影响。前者引起疾病,后者治疗疾病。
在整个人体蛋白质相互作用网络中,受新冠病毒影响的蛋白质形成一个子图,称作疾病模块(Viral Disease Module)。受药物作用的蛋白质形成一个子图,称作治疗模块(Drug Disease Module),见下图。研究两个模块之间的关系是该论文的核心:如果某药物的治疗模块可以作用到疾病模块上或者疾病模块周围,那么该药物很可能有治疗效果。
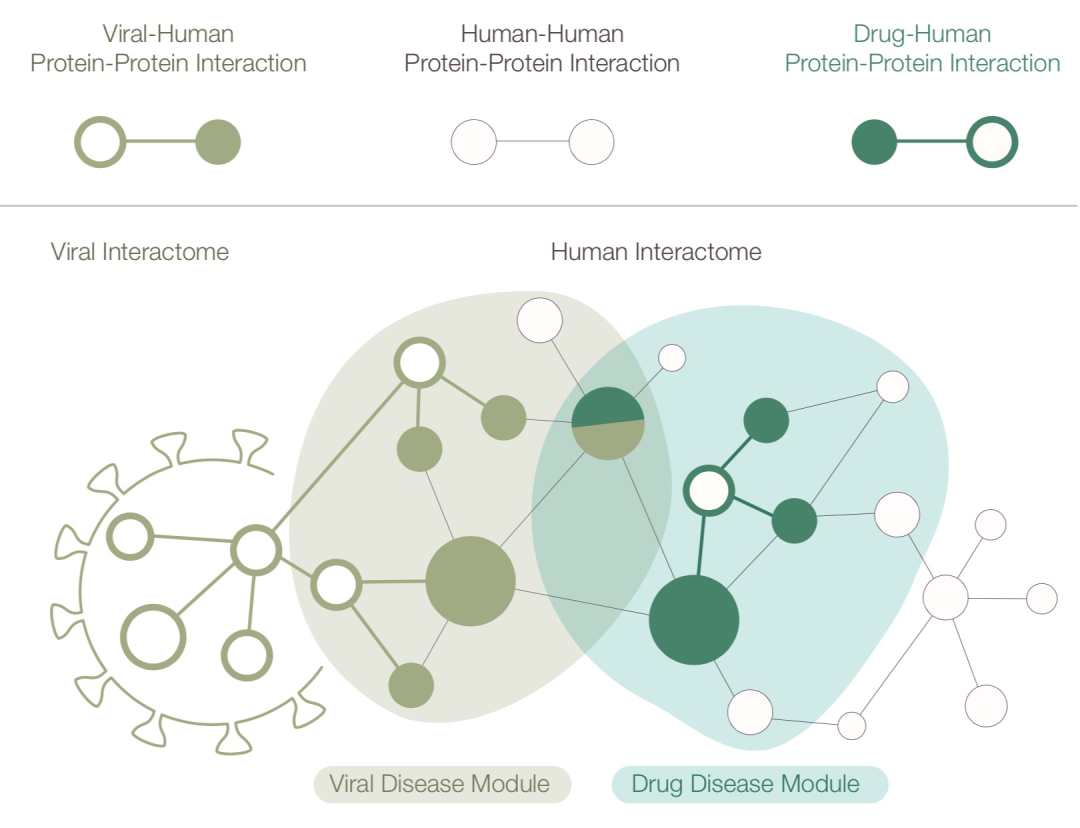
新冠肺炎疾病模块
该论文首先根据已有文献中的结果,选定了 332 个受新冠病毒影响的人体蛋白质。这些蛋白质在人体蛋白质相互作用网络中对应的子图为新冠肺炎疾病模块,见下图。疾病模块中的节点在整个人体蛋白质相互作用网络背景中是分散在各处、彼此不相联系呢?还是集中在同一区域、彼此紧密联系呢?答案是后者。
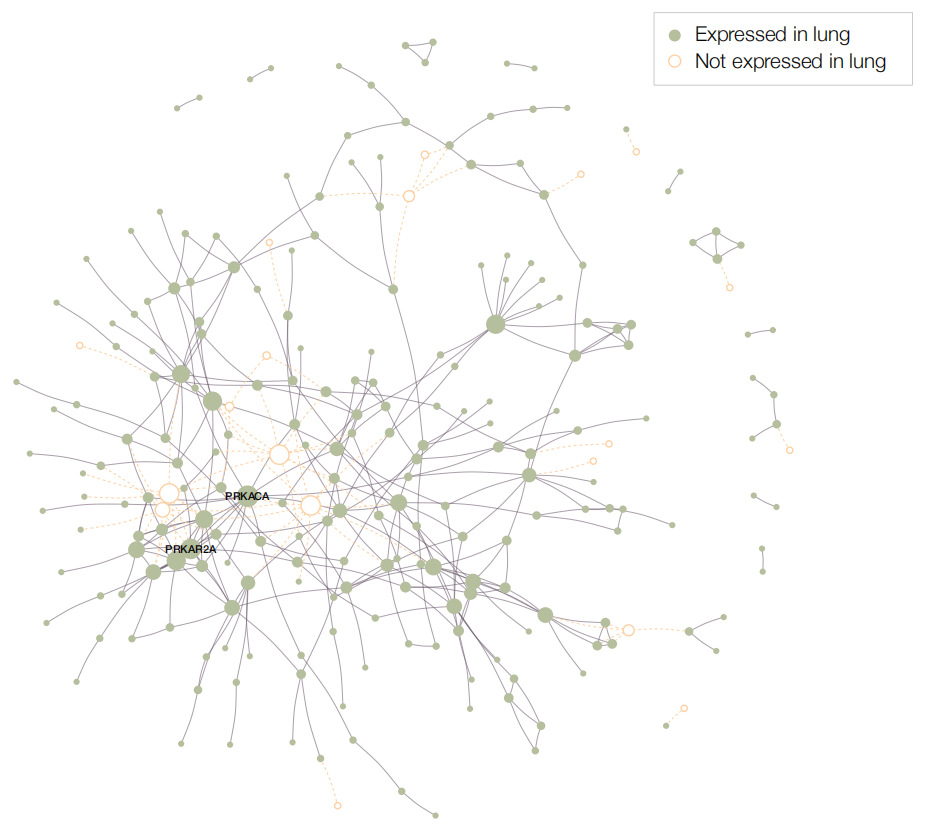
具体研究方法如下。作者计算出疾病模块的最大连通图(Largest Connected Component, LCC)的节点个数为 208,这 208 个节点满足:任意两个节点间有一条路径相连。模块的 LCC 节点数越多,说明模块内部联系越紧密。为了测量 208 个节点是多还是少,作者从人体蛋白质相互作用网络中随机抽取和疾病模块等数量的节点(332个),并保证所选节点与疾病模块中节点的度 (Degree)相同,然后计算LCC节点数。重复此随机过程 1000 次。
结果发现,疾病模块的 LCC 节点数统计上显著地大于随机选取节点的 LCC 节点数。也就是说,疾病模块在人类蛋白质网络中位于一个相对集中的区域。由此得出,治疗模块需要作用于疾病模块内部或周围才能发挥作用。
新冠肺炎与 299 种
疾病之间的相关性
人们在治疗新冠时发现,具有基础疾病的患者更容易发展成重症,预后较差。针对这一现象,作者研究了新冠肺炎与其他疾病之间在生物分子机制上的相关性。理解这种相关性有助于预测新冠引起的并发症。
作者根据已有文献结果,选定每种疾病对应一定数量的致病蛋白,也对应一个疾病模块。然后,将新冠肺炎疾病模块分别与 299 个疾病模块进行比对。结果发现,新冠肺炎疾病模块与其他一些如肺部疾病、心血管疾病、癌症等疾病模块在蛋白质相互作用网络上的距离很近,具有相关性,见下图。此外,新冠肺炎与神经系统疾病在网络上具有相关性,这解释了一些患者在感染初期丧失嗅觉、味觉功能的现象。这些结果在相关文献中得到佐证,并且预测了其他可能发生的并发症。
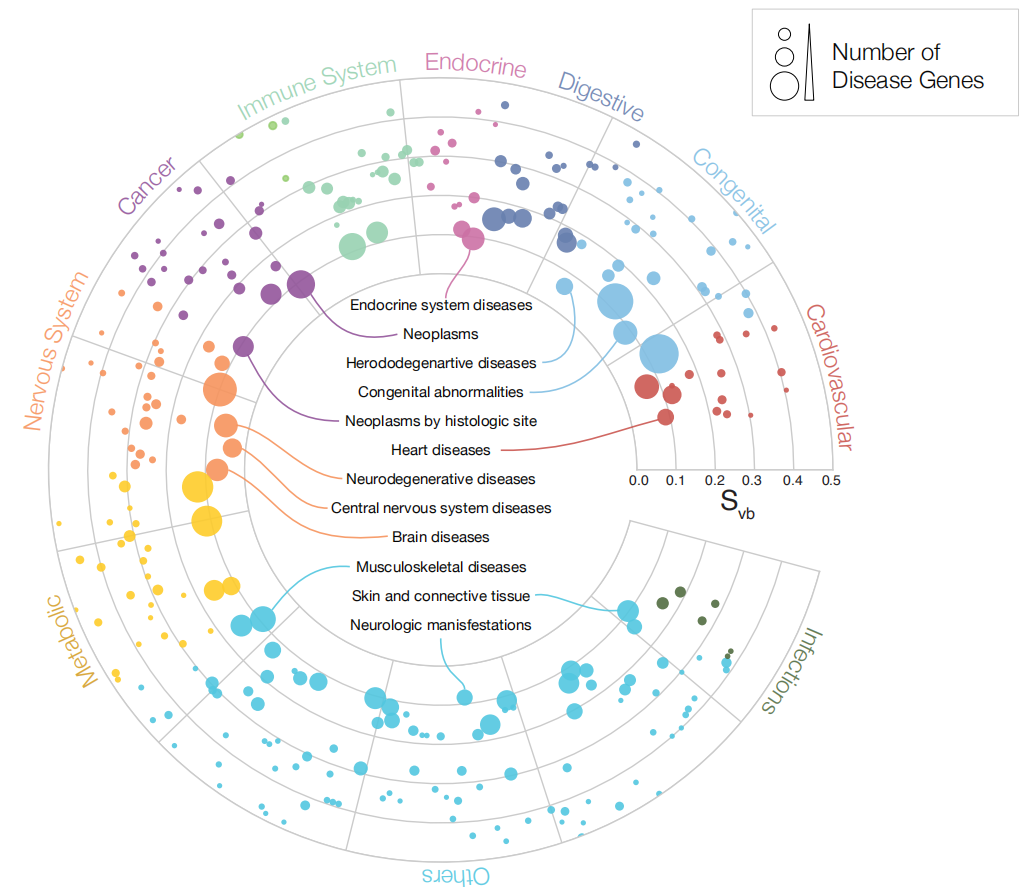
图3:新冠肺炎与 299 种疾病之间的相关性。 圆圈表示疾病,越接近中心的圆圈与新冠肺炎越相关。圆圈大小反映致病蛋白数量,颜色反映疾病所属类型。
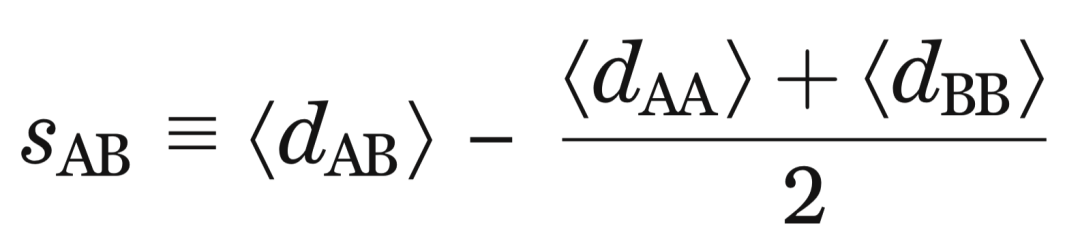
精准预测候选药物
论文的重头戏是预测候选药物。具体来说就是对所有经美国 FDA 批准的药物排名,排名靠前的是有可能对治疗新冠肺炎有效的,列入候选药物。文章给出了包含 81 个候选药物的列表,见下图。有趣的是,在所有 5550 种经美国FDA批准的药物中,引起广泛讨论的“氯喹”排名第五,从侧面证实了预测的准确性。目前,科研团队已根据列表购买候选药物在人体组织细胞中进行试验。对于效果好的化合物后续将进行临床测试。
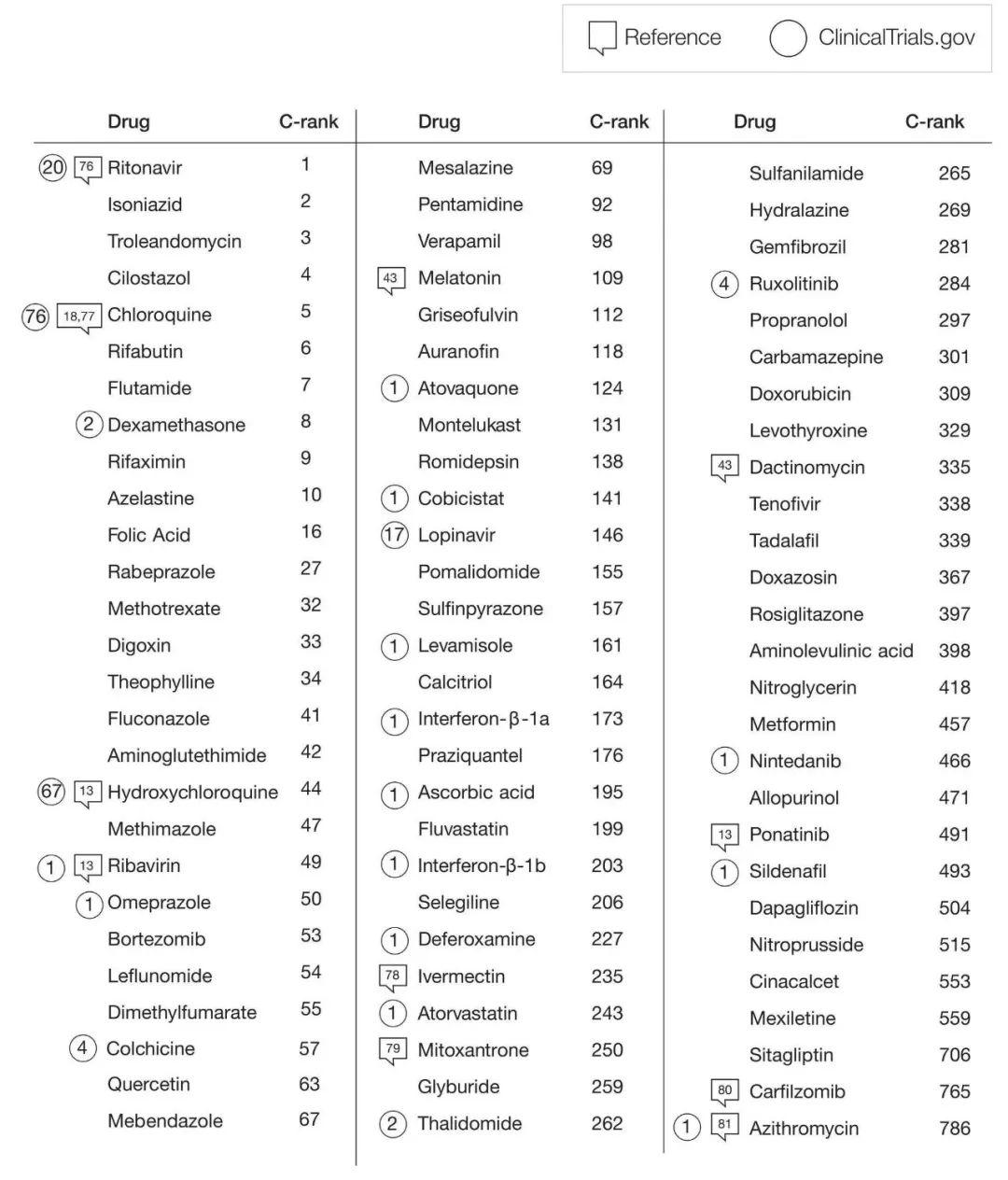
图4:预测得到的候选药物列表。表中包含药物名称和排名,并对一些候选药物标注出了进行临床试验的次数和相关文献佐证。
已有研究发现,在获批准上市的药物中,多数并不直接作用于疾病模块中的蛋白质,而是与模块附近的蛋白质结合。因此,作者认为候选药物的治疗模块不一定直接作用于疾病模块,还可以扰乱疾病模块附近的网络区域。基于这个思路,作者团队采取了三种策略预测候选药物:
-
基于网络距离的策略:候选药物的治疗模块与疾病模块网络距离近,见下图。
-
基于网络扩散的策略:分别扰动疾病模块与治疗模块,通过观察扩散的重叠性来衡量模块间的相似度。候选药物的治疗模块与疾病模块相似度高。
-
基于人工智能的策略:使用图神经网络的方法将疾病与候选药物进行对应。
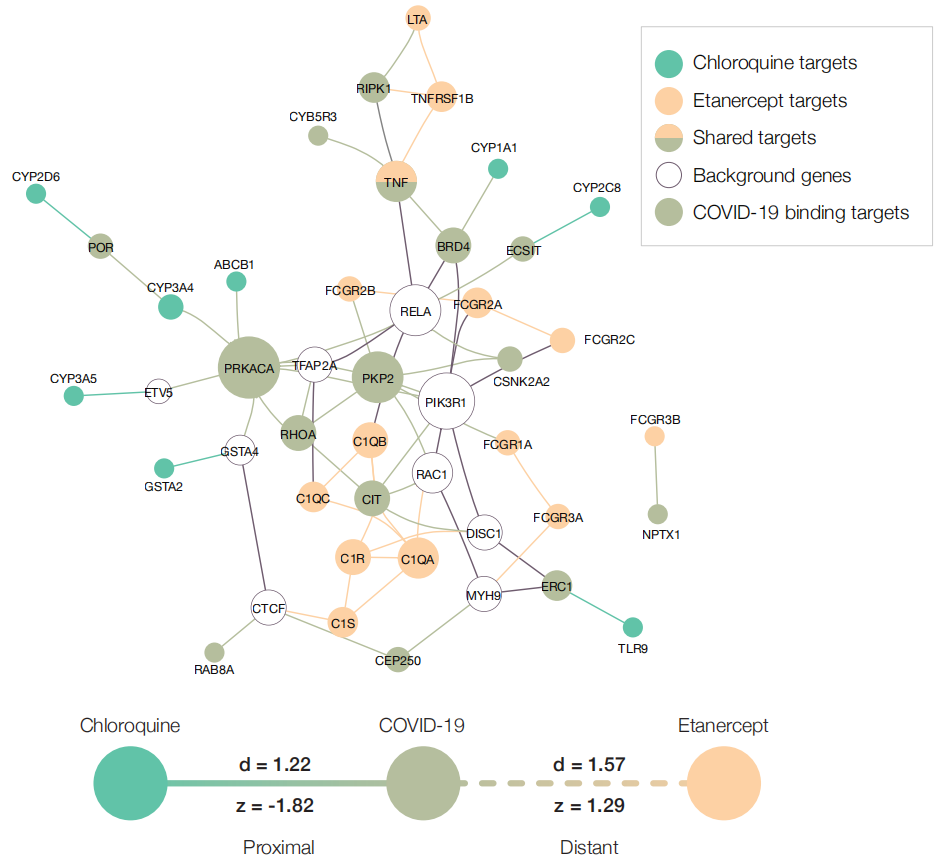
图5:新冠肺炎疾病模块(灰色)与治疗模块之间在网络上的距离示意图。绿色为氯喹治疗模块,橙色为依那西普(治疗关节炎药物)治疗模块。
随后,作者将三种策略分别预测出的结果通过排名聚合算法(Rank Aggregation Algorithm)进行融合,得到最终结果。为了评估预测结果的准确性,作者将预测结果与已知正在进行临床试验的 67 种药物作对比,发现这 67 种药物排名靠前,预测模型的 AUROC 高达 0.89(AUROC 取值在 0.5 到 1 之间,越接近 1 越准确)。此外,作者还发现合并后的结果较三个子模型的结果更为准确。
这篇文章从酝酿到公布仅用时两个月左右。这份成果来源于新冠疫情的紧迫性和网络科学家们的责任感。文末提到:“虽然论文中的方法仍有可继续改进之处,但是鉴于新冠疫情的紧迫性、生物验证的需求以及预测模型的高准确度,我们觉得需要及时披露研究结果,为后续的生物试验与临床测试提供理论基础和指导。”
网络医学相关文献:
[1] Albert-László Barabási, Natali Gulbahce & Joseph Loscalzo. Network medicine:a network-based approach to human disease
[2] Jörg Menche,Amitabh Sharma,Maksim Kitsak, Susan Dina Ghiassian,Marc Vidal,Joseph Loscalzo,Albert-László Barabási. Uncovering disease-disease relationships through the incomplete interactome
[3] Feixiong Cheng,Rishi J. Desai,Diane E. Handy,Ruisheng Wang,Sebastian Schneeweiss,Albert-László Barabási & Joseph Loscalzo-Show fewer authors. Network-based approach to prediction and population-based validation of in silico drug repurposing
作者:刘世康
审校:刘培源
编辑:张爽
推荐阅读
哈佛学者Science刊文:社交疏离措施可能要保持到2022年
Science 最新:旅行控制怎样影响限制新冠病毒的爆发速度?

集智俱乐部QQ群|877391004
商务合作及投稿转载|swarma@swarma.org
◆ ◆ ◆
搜索公众号:集智俱乐部
加入“没有围墙的研究所”

让苹果砸得更猛烈些吧!
👇点击“阅读原文”,了解更多论文信息










