1 克 DNA 可以存储的数据多达 2.15 亿 GB,如果储存得当,DNA 也非常稳定。并有望成为一种极为紧凑稳定的存储介质,但难点在于将计算机及数据构成的数字世界与遗传学的生化世界连接起来。目前,这还依赖于实验室合成 DNA,虽然该方法的成本正在迅速下降,但目前仍然复杂且昂贵。现在,有一种新方法可以使人类将数据信息直接写入活细胞基因组中,实现碳基生物和硅基的连接。

论文题目:
Robust direct digital-to-biological data storage in living cells
论文地址:
https://www.nature.com/articles/s41589-020-00711-4
现代世界产生的数据日益增多,研究人员需要尽快找到可以存储全部数据的新方法。
据估算,到 2025 年,全球每天产生的数据量将达 463 艾字节。
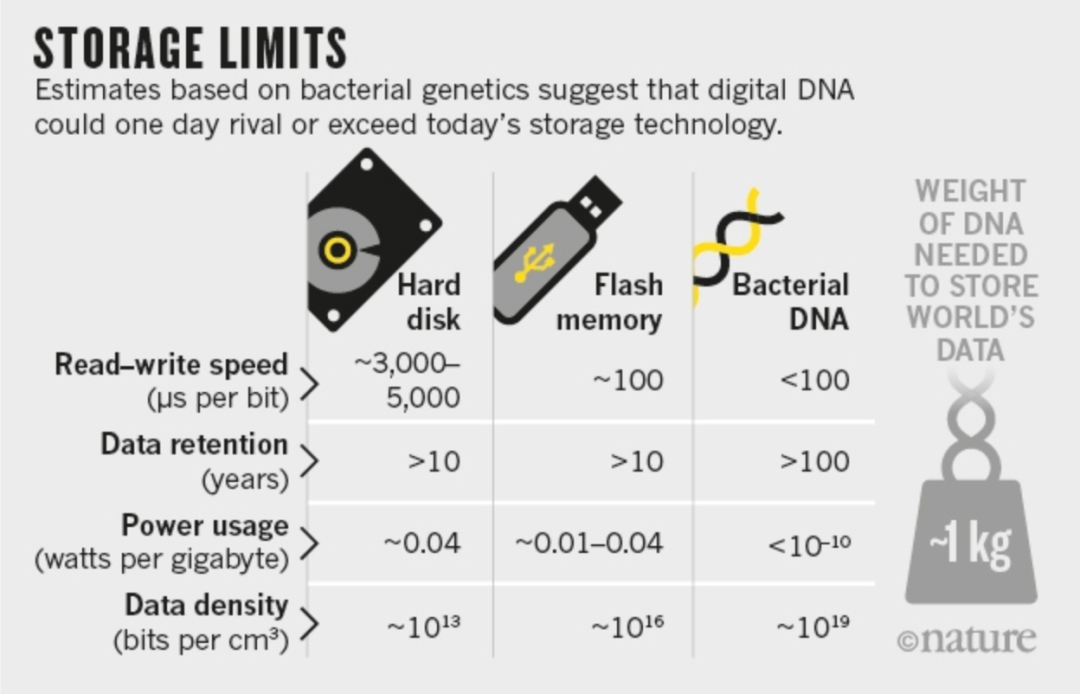
继续采用传统硅技术存储所有数据很快会变得不切实际,但 DNA 可以解决这个问题。首先,DNA 的信息密度比传统硬盘要好上数百万倍,1 克 DNA 可以存储的数据多达 2.15 亿 GB。
不仅如此,如果储存得当,DNA 也非常稳定。2017 年,研究人员仍可以从一匹 70 万年前灭绝的马中提取出完整的基因组。学习使用与自然相同的语言存储和处理数据,同样也可以为生物技术的许多新功能打开大门。
DNA 有望成为一种极为紧凑稳定的存储介质,但难点在于将计算机及数据构成的数字世界与遗传学的生化世界连接起来。
目前,这还依赖于实验室合成 DNA,虽然该方法的成本正在迅速下降,但目前仍然复杂且昂贵。一旦合成完成,在预备使用前,所产生的序列必须在体外小心存放或用 CRISPR 基因编辑技术将其拼接为活细胞。
现在,有一种新方法可以使人类将数据信息直接写入活细胞基因组中,实现碳基生物和硅基的连接:
哥伦比亚大学的研究人员展示了一种新方法 ——“体内电刺激数据记录”(Data Recording in Vivo by Electrical Stimulation,DRIVES),巧妙融合了 DNA 与电子技术,通过电信号调节细胞中的氧化还原生物分子和传感器,直接将数字数据从计算机传输到活细胞中。
该研究刊载于《自然化学生物学》(Nature Chemical Biology),主导研究的 Harris Wang 说,这会引发很多数据存储及更高层次的新应用。
“想象一下,有个可以实时计算和物理重构的蜂窝硬盘驱动器。我们认为,首先要直接将二进制数据编码到细胞中,无需再行 DNA 体外合成”,他说。
“这也许是所有 DNA 存储方法中最困难的部分。如果你能让这些细胞直接与电脑对话,并将基于 DNA 的存储系统与基于硅的存储系统连接起来,那么未来就会有很多可能。”
电子与生物体直接进行信息交换,可以改变我们分析、存储和交流信息的能力。作为主要的生物信息存储介质,DNA 强大的存储能力及稳定性使其逐步走上了数据存储的 “新舞台”。
过去十年中,研究人员在 DNA 数据存储技术上已经取得了一些进展:物理隔离和选择性访问部分数据的存储策略以及优化数据编码及检索的算法进步,极大地提高了 DNA 信息存储的可扩展性和实用性;加之 CRISPR 和重组酶技术的不断进步,DNA 作为高密度数据存储介质的前景广阔。
然而,由于目前人类在体内将数字数据直接编码到活细胞的染色体中的能力仍然有限,目前绝大多数基于 DNA 的数据存储方法仍需要依赖于体外 DNA 完成。
此项工作脱胎自 Wang 先前为大肠杆菌设计的基于 CRISPR 的细胞记录系统,该系统可检测出细胞内的某些 DNA 序列并将其信号记录到生物体的基因组中。
这个系统包括一个基于 DNA 的 “传感模块”,此模块可响应特定的生物信号从而产生高水平的 “触发序列”,记录仪的 “DNA 传真条(DNA ticker tape)” 将这些序列整合起来,最终完成信号记录。
在新的进展中,Wang 及其同事对传感模块进行了改装,使其可与另一研究电信号反应的团队所开发的生物传感器配合使用。他们将大量细菌放置在一个由一系列小室( chambers )组成的设备中,这些小室使研究小组能够将试验细菌暴露于电信号下。
研究建立在之前研发的定向 DNA 写入系统基础上。该系统可以利用 CRISPR 间隔子采集技术,检测出细胞内的某些 DNA 序列并将其信号记录到生物体的基因组中。而在新研究中,研究团队在原有系统上增加了写入信息的方向性功能以及用于缩放的临时记录和多路复用功能。
当通电时,触发序列的水平上升并录入 DNA 传真条。具有高比例触发序列的片段表示二进制的 “1”,而缺失则表示 “0”,这使得研究人员可以直接将数字信息编码到细菌的基因组中。
单个细胞可以容纳的数据量非常小,只有 3 位。因此研究人员设计了一种方法,用不同的 3 位数据块同时编码 24 个单独细菌种群,共计可得 72 位。
他们用此方式将 “hello world!(意为你好,世界!)” 这一信息编入细菌,并通过对组合群体进行测序及使用专门设计的分类器将显示该信息,检索信息准确率可达 98%。
值得注意的是,DNA 在复制过程中存在突变。虽然经过此前实验验证,存储数据的 CRISPR 阵列中的间隔子在 50 代以上是稳定的。然而,随着时间的推移,Cas1–Cas2 复合体会出现细微变化,并逐渐放大到可能影响数据恢复的水平。
为此,研究人员进行继续进行实验,在 80 代的种群中,数据解码准确率仍在 90% 以上。这表明,活细胞中编码的 72 位数据可以在 80 次迭代中以指数和自主方式放大,以产生约~1.2 × 1024 (280) 倍仍然可以被可靠地解码物理拷贝。
DNA 的稳定性和可访问性是其存储数据的关键优势。
不过,在体外的开放自然环境中,存在诸多如 DNA 酶、微生物、紫外线和化学诱变剂等因素,影响对 DNA 的数字信息的保存,但活细胞可以很好解决这一难题。
为了研究自然环境中存储在细胞中的数据的完整性,研究人员将一个载有 54 字节信息的细胞群放入未灭菌的盆栽土中,然后对该土壤微生物群落进行测序,仍能检索到高达 90% 的数据。
此外,采用较低突变率的工程宿主菌株或其他生物控制策略、减少氧化还原转换电子信号引起的批间变异、冻干或使用孢子形成菌也会提高活细胞内的数据保真度。
这一打通 “数字” 与 “生物” 的数据存储策略,同样也可以应用于其他具有独特性质(如天然电活性、快速生长或极端耐受性)的微生物系统。
使用 DNA 存储数据是提供更高密度存储的关键研究领域。
显然,72 位距离现代硬盘的存储容量还有很长的路要走,甚至无细胞的 DNA 存储技术现在也只以千兆字节为单位。
但 Wang 表示,这仅仅是此观念的证明,为记录仪供电的 CRISPR 机械效率、能够可靠读取的 “传真条” 长度、甚至用于编码数据的电子设备都有很大提升空间。
“所有这些在未来几年内都会有所改善,我坚信,即使在短期内,也有将系统容量大规模扩展几个数量级的可能。”Wang 说。
此外,他还补充道,将数据储存在细胞而非体外的优势明显。首先,放大或复制数据会便宜得多,只要简单地多培养些细胞就可以,不必再进行复杂的人工 DNA 合成。研究小组在论文中表示,所记录的信息在 60 到 80 代细胞中保持稳定。
其次,细胞天然具有使 DNA 免受环境干扰的能力。为证明这一点,研究人员将大肠杆菌细胞放入未灭菌的盆栽土中,然后对该土壤微生物群落进行测序,能够确实检索到一个 52 位的信息。
最令人兴奋的也许是,将这种数据记录能力与生物计算机新兴研究相结合的可能性。
虽然目前已有研究者开始对细胞的 DNA 进行改造,使其能够执行逻辑和存储操作,但在硅和基因组之间建立直接接口,可以显着提高我们自己的设备对细胞进行重新编程的能力。
最后值得一提的是,早在 2019 年 3 月,微软和美国华盛顿大学展示了一种用于存储 DNA 数据的自动化流程,但这使用了人工合成的 DNA。它以前曾设法在 DNA 上存储 1Gbit 的数据,但自 2019 年底以来,相关团队似乎并未公布更多进展。
集智斑图顶刊论文速递栏目上线以来,持续收录来自Nature、Science等顶刊的最新论文,追踪复杂系统、网络科学、计算社会科学等领域的前沿进展。现在正式推出订阅功能,每周通过微信服务号「集智斑图」推送论文信息。扫描下方二维码即可一键订阅:
点击“阅读原文”,追踪复杂科学顶刊论文