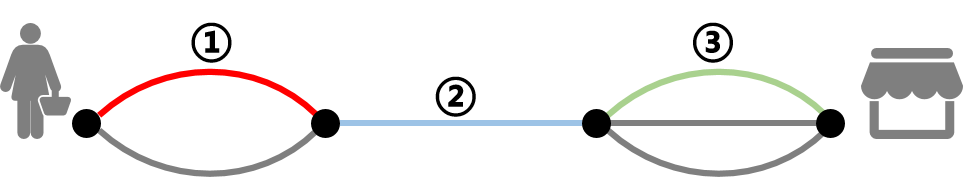虽然在生活中我们总会说每个人都是平等的,但是在某些特定领域或者问题上,我们必须要承认某些人更为重要。这就出现了“人人平等”,但有些人“更为平等”的有趣现象。该现象在生化网络中又如何呢?近日发表在 PNAS 上的一篇文章为了更好地捕捉生化调控和信号传导的功能相关信息,从而引入有效图(effective graph)的概念来更好地用于生化调控、信号传递等。针对这项研究,PNAS 评论文章从有效图必要性、创新性和生化应用等方面进行解读,并引发读者关于生化网络连边平等、冗余及交互的思考。
Kishore Hari, Uday Ram, Mohit Kumar Jolly | 作者
胡一冰 | 译者
刘培源 | 审校
邓一雪 | 编辑
生物系统经常会执行多种任务,而它们若想有效地完成这些任务时,它们往往面临着多重权衡。因此,生物系统及其组成部分会通过大量的试错来达到最优[1]。这种优化类似于人类制造的机器通过反复试错来提升此类工程系统的效率。然而,工程系统的运作也需要考虑资源和成本效益,也就是说,工程师会主动从一台机器上拆卸与指定任务无关的部件。那么在生物系统中也会进行类似的“减负”吗?针对这个问题,近日在 PNAS 中,美国东北大学 Alexander J.Gates 等研究者们[2]给出了否定答案。他们分析了生物系统中的多种调节网络(Regulatory networks),并发现所有这些网络都包含冗余的相互作用,而这些相互作用并不直接促进它们执行的细胞功能。
生物系统中普遍存在着多种规模大小和复杂程度的调节网络。这些网络中又蕴含着各种生物化学分子之间复杂的相互作用。细胞的功能,例如细胞分化和细胞周期调节,都来自于这样存在复杂相互作用关系的网络。最近的高通量(high throughput)技术的进步有利于全面表征分子相互作用的各种细胞过程,揭示相互依存的复杂网络关系[3]。这种海量数据引发概念性问题——这些大规模的交互作用有什么用,以及网络中所有的连边是否在功能性上重要程度都是平等的,但有些边比其他边更加平等。
图1. 《动物庄园》是英国作家乔治·奥威尔于1945年出版的动物寓言题材小说,该作被公认为反乌托邦政治讽喻寓言。该书中的经典名句“All animals are equal, remember. But some animals are more equal than others.”(所有的动物一律平等,但有的动物比其他动物更平等)。
目前有很多种计算方法被开发并应用来捕捉调节网络紧急动态的关键特征。这些模型通常旨在基于一套用数学算式表达、反映网络节点间相互作用的规则。这类模型中最简单的一类是布尔网络(Boolean networks, BNs)模型[4]。顾名思义,在这种模型中,网络中一个节点的值假设只有两个状态:0(低)和1(高)。这些规则由可利用的实验文献结合而成,这些实验文献涉及所有影响给定节点的边的组合效应,这些假设要么弥补了生物信息的不足,要么有助于降低数学复杂性[5]。尽管基于这些简单的假设,布尔网络在半个多世纪以来成功地应用于描述和预测动态属性,例如显性表征和细胞周期控制等[6-7]。然而,布尔网络中所有边都具有同等的重要性,而对于特定的网络拓扑结构,某些边对网络涌现个别属性的贡献程度可能存在显著差异。生物网络对许多结构或动力扰动非常鲁棒,但对某些扰动高度敏感,因此不同的状态(表现型)应运而生。换句话说,生物网络中包含某些边比其他边“更为平等”,但目前布尔网络建模策略并没有明确考虑网络相互作用中冗余的必要性和影响效力。
图2. 外出购物的路径示意图。若②路段失效,则居民无法抵达商场,它是不可替代的;若①或③路段失效,居民仍可以选择替代路段前进,但考虑对应的替代路段数量,边不可替代程度①>③。本质上看,所有路段都会承载一定的服务需求,所有边都是“平等”的。但从完成购物目的角度来看,②比①和③“更为平等”。
冗余可以通过观察生物化学网络中“渠化”(canalization)的存在来证实[10],只要对网络进行少量的动态扰动,就可以改变涌现的网络特性[11]。此外,小型网络结构[12-14]就能够执行多种动力学行为,如表现型切换、适应、脉冲产生和振荡,这表明大型网络的能力存在冗余。那我们如何理解这种冗余的程度,并将其应用在我们理解生物网络并对其进行抗扰预测的研究中去呢?
图3. 有效图识别网络相互作用中的冗余。(A)三个节点、六个边的网络样本。(B)对应的有效图,数值表示每六条边的有效性得分。(C)查找A中网络对应的真值表。(D)表征边对节点A和C的有效性。#(通配符)表示节点的活动,不影响网络输出。
Gates等人[2]提出了一种量化布尔网络中这种冗余的方法。简单地说,他们的方法称为“有效图”(effective graph),使用布尔网络查真值表(如图1)来量化输入节点的活动(以及相应的边沿)对相应目标节点状态的影响。他们为从一个起点到一个终点的每条边打分——称为“边有效性”,分值从0到1,其中0表示完全多余的边(因此可以在不干扰网络动力学的情况下删除),1是必须存在的边。因此,有效图可以知道在某个网络某条边是否是完全或几乎完全冗余,也可以用于识别模块结构(一组节点在网络中相互联系紧密,但相对其他节点联系较弱),还解释扰动如何从某个节点(比如药物治疗)渗透进全网络,最终影响细胞功能。
通过这种方法,研究者们将大量不同规模和复杂程度的布尔网路中边的有效性属性化,并发现所有这些网络至少有一个完全冗余的边。例如,删除至少一个边不会干扰网络上的动力学或生物功能。有趣的是,研究者们观察到,这些布尔网络边的平均有效度明显小于随机构建的“假设”网络(即平均冗余程度更大)。这些观察表明,冗余在生物网络中是普遍存在的,并且可能已经被进化所选择。该项目提出了这种布尔网络的结构冗余作为一种疏导机制,只有有限的扰动集(针对网络中的特定边或节点)才能改变网络动态。它们有力地证明了有效图比整个交互图(interaction graph)能更好地预测扰动的结果。因此,有效图可能在识别调节网络的“阿基里斯之踵”方面提供更多信息。这让人不禁想起另一种网络简化方法,通过得到特定网络的“影响矩阵”(influence matrix)来衡量各个节点之间的作用程度[15]。
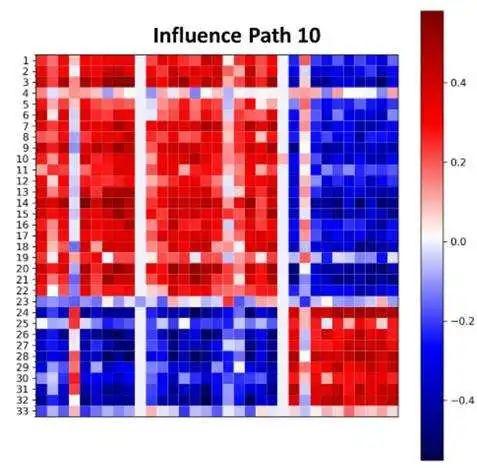
图4. “影响矩阵”,可用来量化节点对其他节点的影响,取值范围从-1到1,详见参考文献[15]。
Gates 等人[2]提出的有效图在概念上与代谢网络中观察到的功能冗余相似,代谢通量(metabolic flux)可以利用网络中的许多路径。这样的冗余使得新陈代谢对许多致命的干扰都很鲁棒[16]。利用有效图,作者建议识别出最能代表从无数输入信号到有限输出表现型的信息流的控制路径。这些路径可以引导我们尝试确定病理情况下,能破坏潜在的控制结构治疗策略。
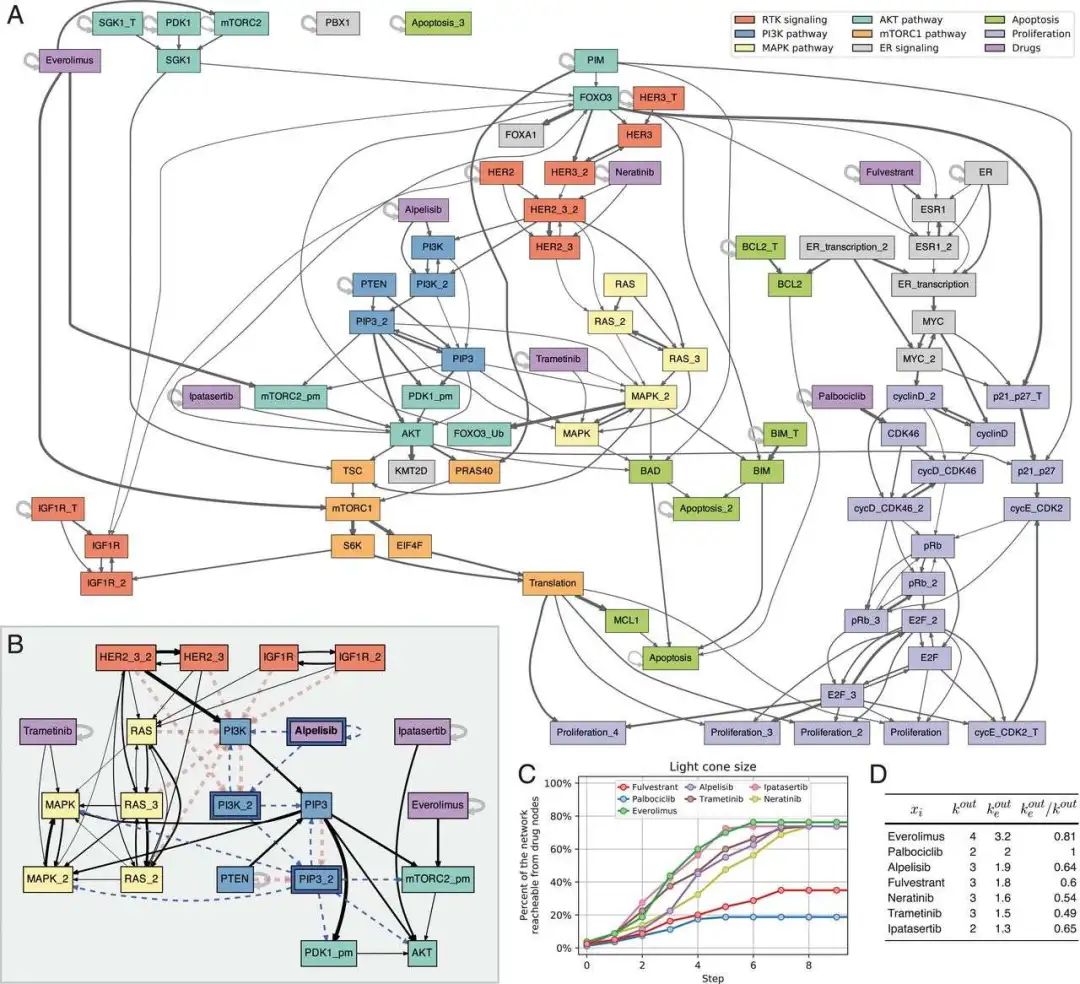
通过使用潜在的信号网络决定细胞是否会发生凋亡或增殖的ER+乳腺癌案例研究证明了该方法的有效性。对冗余边的识别和删除导致网络被分割成不同的模块。这些模块不仅识别了网络中的各种控制路径,而且还表明,从信号到节点的可达性远低于交互图识别的可达性。也就是说,个体扰动在影响细胞动力学方面可能不像默认假设的那么有效。这些对网络拓扑结构的洞见揭示了许多路径表现出对致命干扰的承受力,至少许多致命结果是因为子模块分裂导致。因此可知,实际中需要多种治疗扰动来减少癌症的数量。
图5. 针对ER+乳腺癌BN模型的研究。A为ER+乳腺癌的BN模型有效图的分层绘制,B为条件有效图,C为扰动对模型中7种药物各自的扩散动力学和网络有效可达比例,D为药物变量出边的有效性
考虑到所开发方法的可扩展性,该方法可用于识别有效特征,它可以辅助各种网络还原策略,这些策略通常以保留网络的紧急特性为目标,最常见的策略是网络启用的吸引子(或表现型)[17]。有效图基于查找真值表的结果对网络进行简化,保证了吸引子的守恒性。此外,它还提供了关于简化网络的连通程度的信息。虽然简化的网络表示方法,包括有效图,已经能够解释各种实验现象,但它们实现非直观或非琐碎的预测的能力并达到预期结果(例如减少癌细胞数量)取决于组合或顺序的干扰。
从概念上讲,有效图法非常有用,应该可以被广泛使用。然而,我们需要对一些固有假设保持谨慎。有效图的推理完全依赖于用于开发上述布尔网络的规则,而这些规则会随着不断增加的实验要求而改变。这种依赖性可能会阻碍这些结果的推广,但是在生物系统中重复的渠化疏导可以解决这些问题。此外,定义特定细胞系或环境特异性有效图,对类似于试图定义癌与肉瘤的调控网络[18]可能会有所帮助。
一个重要的概念性问题在此仍有待回答:是什么机制导致了如此广泛的冗余?多种因素可能会有影响,例如(1)基于实验的网络管理错误;(2)在进化时间尺度上的网络剪枝“反复试验”以及(3)这些冗余部分具有迄今未被识别的功能。无论原因是什么,要回答为什么生物网络中的某些连边可能“比其他连边更平等”,这绝对是一段有趣的探索之旅。
https://www.pnas.org/content/118/16/e2103698118
[1] O. Shoval et al., Evolutionary trade-offs, Pareto optimality, and the geometry of phenotype space. Science 336, 1157–1160 (2012).
[2] A. J. Gates, R. Brattig Correia, X. Wang, L. M. Rocha, The effective graph reveals redundancy, canalization, and control pathways in biochemical regulation and signaling.Proc. Natl. Acad. Sci. U.S.A.118,e2022598118 (2021).
[3] M. M. Saint-Antoine, A. Singh, Network inference in systems biology: Recent developments, challenges, and applications. Curr. Opin. Biotechnol. 63, 8 9–98
[4] J. D. Schwab, S. D. Kühlwein, N. Ikonomi, M. Kühl, H. A. Kestler, Concepts in Boolean network modeling: What do they all mean? Comput. Struct. Biotechnol. J.18, 571–582 (2020).
[5] F. Font-Clos, S. Zapperi, C. A. M. La Porta, Topography of epithelial mesenchymal plasticity. Proc. Natl. Acad. Sci. U.S.A. 115, 5902–5907 (2018).
[6] R. S. Wang, A. Saadatpour, R. Albert, Boolean modeling in systems biology: An overview of methodology and applications. Phys. Biol. 9, 055001 (2012).
[7] S. Bornholdt, Boolean network models of cellular regulation: Prospects and limitations. J. R. Soc. Interface 5 (suppl. 1), S85–S94 (2008).
[8] K. Hari et al., Identifying inhibitors of epithelial-mesenchymal plasticity using a network topology-based approach. NPJ Syst. Biol. Appl. 6, 15 (2020).
[9] R. B. Correia, A. J. Gates, X. Wang, L. M. Rocha, CANA: A Python package for quantifying control and canalization in Boolean Networks. Front. Physiol. 9, 1046
[10] C. H. Waddington, Canalization of development and genetic assimilation of acquired characters. Nature 183, 1654–1655 (1959).
[11] M. L. Siegal, A. Bergman, Waddington’s canalization revisited: Developmental stability and evolution. Proc. Natl. Acad. Sci. U.S.A. 99, 10528–10532 (2002).
[12] U. Alon, Network motifs: Theory and experimental approaches. Nat. Rev. Genet. 8, 450–461 (2007).
[13] D. Jia et al., Quantifying cancer epithelial-mesenchymal plasticity and its association with stemness and immune response. J. Clin. Med. 8, 725 (2019).
[14] W. Ma, A. Trusina, H. El-Samad, W. A. Lim, C. Tang, Defining network topologies that can achieve biochemical adaptation. Cell 138, 760–773 (2009).
[15] L. Chauhan, U. Ram, K. Hari, M. K. Jolly, Topological signatures in regulatory network enable phenotypic heterogeneity in small cell lung cancer. Elife 10,10.7554/eLife.64522 (2021).
[16] G. Sambamoorthy, K. Raman, Understanding the evolution of functional redundancy in metabolic networks. Bioinformatics 34, i981–i987 (2018).
[17] J. G. T. Za~nudo, R. Albert, An effective network reduction approach to find the dynamical repertoire of discrete dynamic networks. Chaos 23, 025111(2013).
[18] J. A. Somarelli et al., Mesenchymal-epithelial transition in sarcomas is controlled by the combinatorial expression of microRNA 200s and GRHL2. Mol. Cell. Biol.36, 2503–2513 (2016).
集智学园特邀陈关荣、项林英、樊瑛、宣琦、李翔、史定华、李聪、荣智海、周进、王琳等网络科学专家作为导师,依托汪小帆、李翔、陈关荣的经典教材《网络科学导论》,自2月27日起开展系列上线课程,以网络动力学为主线构建网络科学知识体系。欢迎希望进入网络科学领域、提高网络分析能力、与一线专家探讨问题的朋友报名参加!

点击查看课程详情:
点击“阅读原文”,追踪复杂科学顶刊论文