Nat. Mach. Intell. 速递:利用胶囊网络预测复杂遗传病的患病率
关键词:胶囊网络,层级结构,非线性,复杂网络


论文题目:Predicting the prevalence of complex genetic diseases from individual genotype profiles using capsule networks 论文来源:Nature Machine Intelligence 论文链接:https://www.nature.com/articles/s42256-022-00604-2
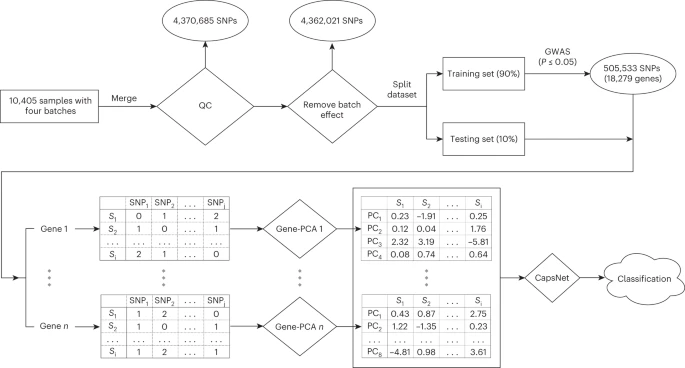
图1. 疾病胶囊的运作流程,基于来自4个批次的10456个全基因数据,经过质控,批次效应去除,选出合适4.3M个SNP,之后分为测试和训练数据集,通过 PCA 降维,将降维后的结果经由胶囊网络得到分类结果。
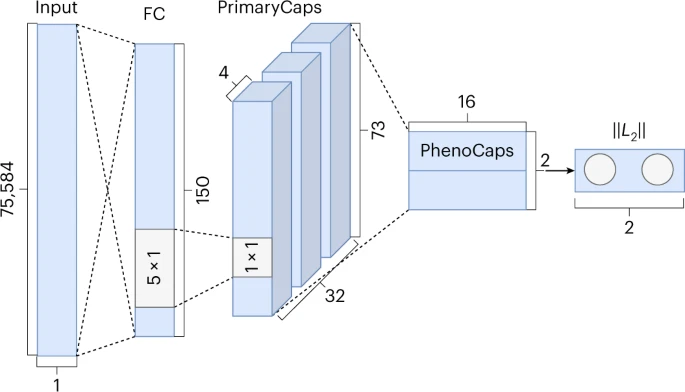
图2. 疾病胶囊的网络架构,输入是来自所有 Gene-PCA 模型的压缩特征向量,其中每个特征对应于一个 Gene-PCA。基因主成分分析的数量为 75,584,因此输入维数为 75,584 × 1。疾病胶囊由三层组成: 完全连接层(FC)、初级胶囊层 (PrimaryCaps) 和表型胶囊层 (phenoCaps)。FC 层由150个神经元组成,其次是激活函数。PrimaryCaps 由32个主胶囊组成。它们每个都包含四个不同的卷积滤波器(内核大小5 × 1,步长2,无填充)。表型胶囊由两个16维向量组成。每个表型胶囊接受来自所有32个初级胶囊的输入。输出是一个二元分类标签(健康或 ALS)。
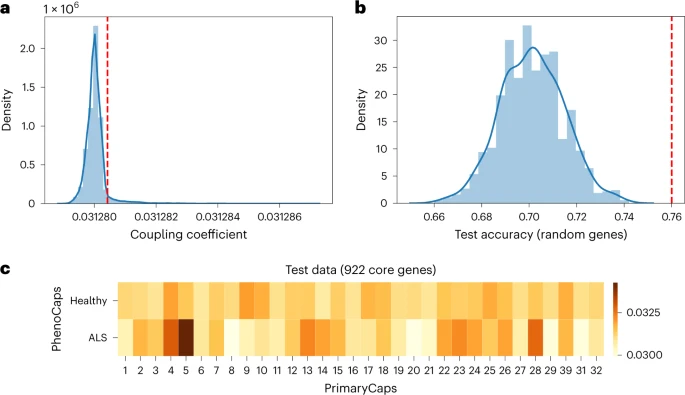
图3. 初级胶囊与表型胶囊在ALS上所有基因中的的偶联系数分布。红色虚线表示95%百分位。以上922个基因作为分类的决定性核心基因。B,使用922个随机选择的基因作为疾病胶囊模型的输入(重复1,000次) ,而其他基因被掩盖(设置为零)的测试准确性分布。红色虚线表示使用922个核心基因作为输入的测试准确性。C,以922个核心基因为输入的平均耦合系数矩阵热图(测试数据)。
高阶网络社区
详情请见:
推荐阅读
-
319篇文献、41页综述文章讲述图神经网络用于医疗诊断的前世今生与未来 -
AI不再黑箱:利用可解释的胶囊网络算法识别细胞亚型 -
捕捉生物调节过程中的时滞现象:非线性微分方程与生物网络建模 -
《张江·复杂科学前沿27讲》完整上线! -
成为集智VIP,解锁全站课程/读书会 -
加入集智,一起复杂!