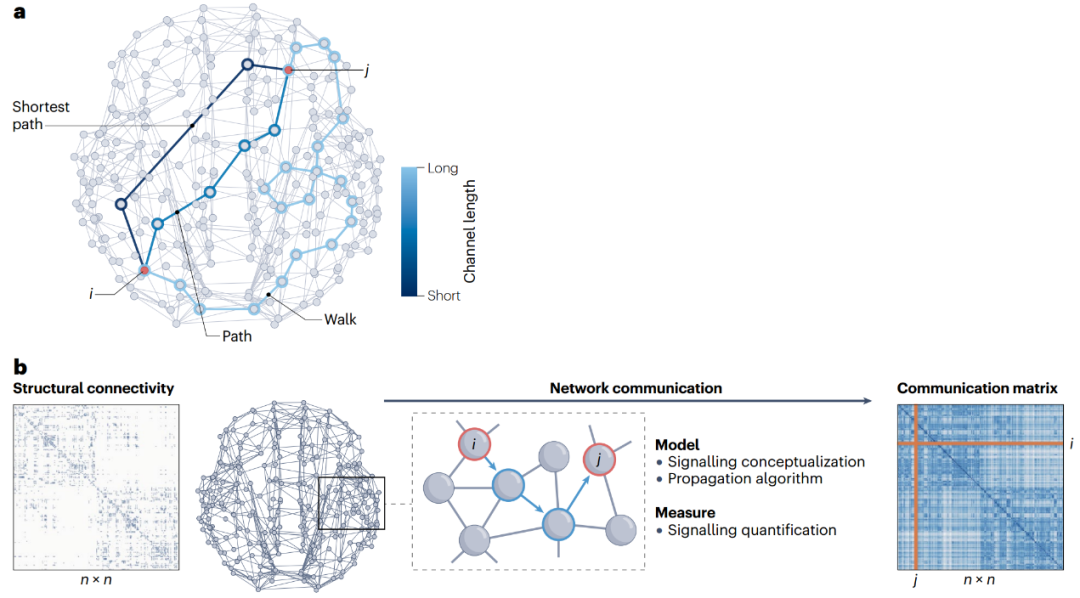
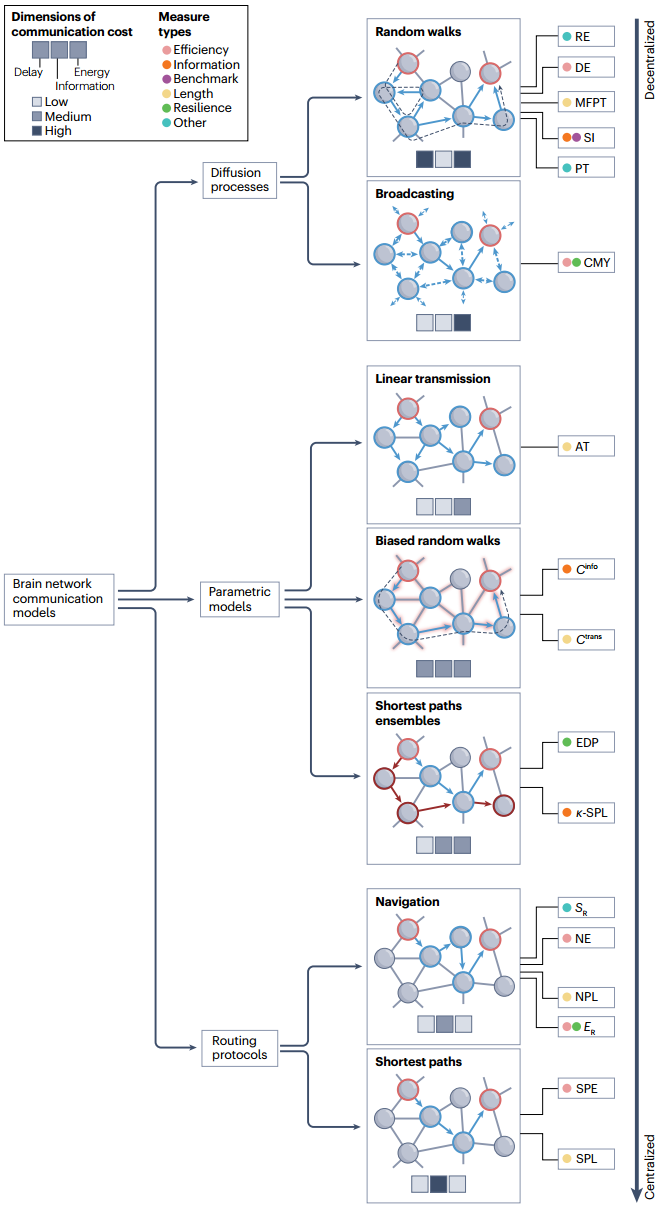
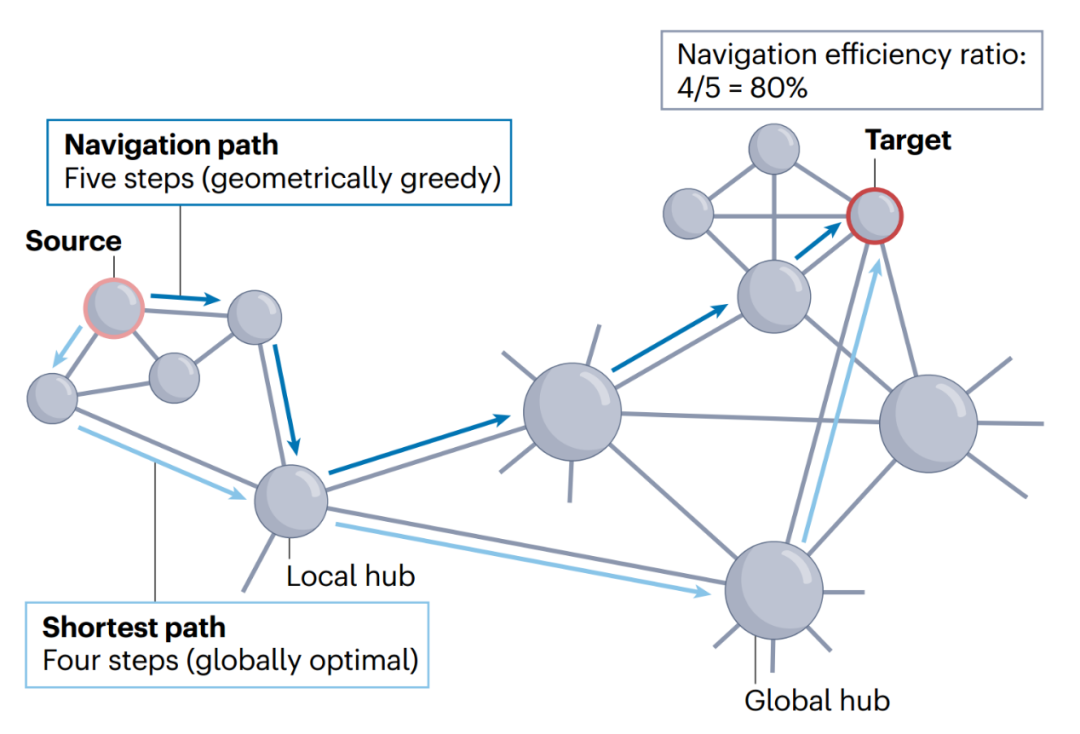
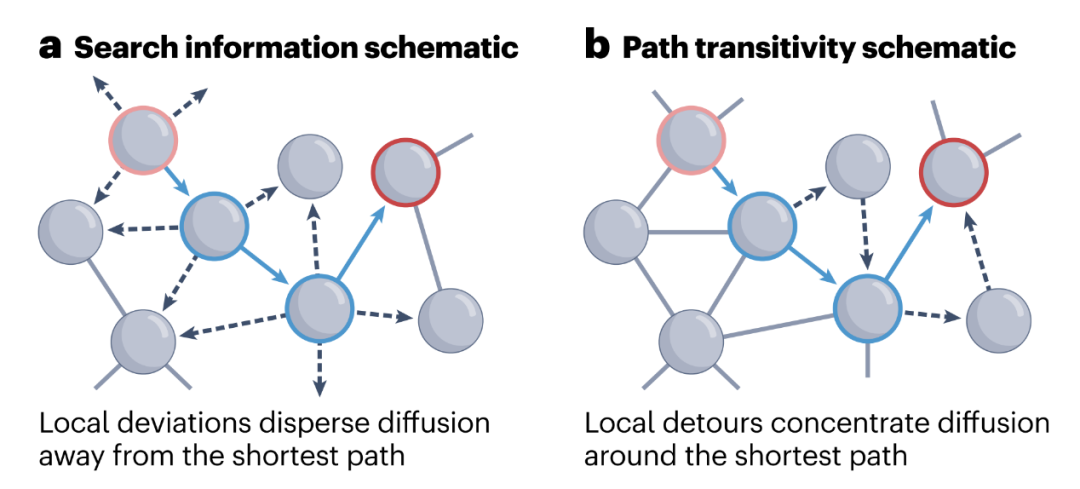
-
Avena-Koenigsberger, A., Misic, B. & Sporns, O. Communication dynamics in complex brain networks. Nat. Rev. Neurosci. 19, 17–33 (2018).
This Review was one of the first proposals of network communication as a conceptual framework to bridge the gap between brain structure and function.
-
Laughlin, S. B. & Sejnowski, T. J. Communication in neuronal networks. Science 301, 1870–1874 (2003).
-
Debanne, D., Bialowas, A. & Rama, S. What are the mechanisms for analogue and digital signalling in the brain? Nat. Rev. Neurosci. 14, 63–69 (2013).
-
Hahn, G., Ponce-Alvarez, A., Deco, G., Aertsen, A. & Kumar, A. Portraits of communication in neuronal networks. Nat. Rev. Neurosci. 20, 117–127 (2019).
This paper reviews putative mechanisms of communication in small networks of neuronal populations.
-
Yeo, B. T. T. et al. The organization of the human cerebral cortex estimated by intrinsic functional connectivity. J. Neurophysiol. 106, 1125–1165 (2011).
-
Kumar, A., Rotter, S. & Aertsen, A. Spiking activity propagation in neuronal networks: reconciling diferent perspectives on neural coding. Nat. Rev. Neurosci. 11, 615–627 (2010).
-
Buzsáki, G. & Wang, X.-J. Mechanisms of gamma oscillations. Annu. Rev. Neurosci. 35, 203–225 (2012).
-
Fries, P. Rhythms for cognition: communication through coherence. Neuron 88, 220–235 (2015).
-
Kaufman, M. T., Churchland, M. M., Ryu, S. I. & Shenoy, K. V. Cortical activity in the null space: permitting preparation without movement. Nat. Neurosci. 17, 440–448 (2014).
-
Semedo, J. D., Zandvakili, A., Machens, C. K., Yu, B. M. & Kohn, A. Cortical areas interact through a communication subspace. Neuron 102, 249–259.e4 (2019).
-
Deco, G., Jirsa, V. K., Robinson, P. A., Breakspear, M. & Friston, K. The dynamic brain: from spiking neurons to neural masses and cortical fields. PLoS Comput. Biol. 4, e1000092 (2008).
-
Palmigiano, A., Geisel, T., Wolf, F. & Battaglia, D. Flexible information routing by transient synchrony. Nat. Neurosci. 20, 1014–1022 (2017).
-
Bassett, D. S. & Sporns, O. Network neuroscience. Nat. Neurosci. 20, 353–364 (2017).
-
Bullmore, E. & Sporns, O. Complex brain networks: graph theoretical analysis of structural and functional systems. Nat. Rev. Neurosci. 10, 186–198 (2009).
-
Fornito, A., Zalesky, A. & Bullmore, E. T. Fundamentals of Brain Network Analysis(Academic, 2016).
This book provides a comprehensive introduction to network neuroscience and connectomics.
-
Javadzadeh, M. & Hofer, S. B. Dynamic causal communication channels between neocortical areas. Neuron 110, 2470–2483.e7 (2022).
-
Park, H.-J. & Friston, K. Structural and functional brain networks: from connections to cognition. Science 342, 1238411 (2013).
-
Suárez, L. E., Markello, R. D., Betzel, R. F. & Misic, B. Linking structure and function in macroscale brain networks. Trends Cogn. Sci. 24, 302–315 (2020).
-
Hagmann, P. et al. Mapping the structural core of human cerebral cortex. PLoS Biol. 6, e159 (2008).
-
van den Heuvel, M. P. & Sporns, O. Rich-club organization of the human connectome. J. Neurosci. 31, 15775–15786 (2011).
-
Meunier, D., Lambiotte, R. & Bullmore, E. T. Modular and hierarchically modular organization of brain networks. Front. Neurosci. 4, 200 (2010).
-
Bassett, D. S. & Bullmore, E. Small-world brain networks. Neuroscientist 12, 512–523 (2006).
-
van den Heuvel, M. P., Bullmore, E. T. & Sporns, O. Comparative connectomics. Trends Cogn. Sci. 20, 345–361 (2016).
-
Bullmore, E. & Sporns, O. The economy of brain network organization. Nat. Rev. Neurosci.13, 336–349 (2012).
-
van den Heuvel, M. P., Kahn, R. S., Goñi, J. & Sporns, O. High-cost, high-capacity backbone for global brain communication. Proc. Natl Acad. Sci. USA 109, 11372–11377 (2012).
-
Cole, M. W., Ito, T., Bassett, D. S. & Schultz, D. H. Activity flow over resting-state networks shapes cognitive task activations. Nat. Neurosci. 19, 1718–1726 (2016).
-
Shine, J. M. et al. Human cognition involves the dynamic integration of neural activity and neuromodulatory systems. Nat. Neurosci. 22, 289–296 (2019).
-
van den Heuvel, M. P. & Sporns, O. A cross-disorder connectome landscape of brain dysconnectivity. Nat. Rev. Neurosci. 20, 435–446 (2019).
-
Fornito, A., Zalesky, A. & Breakspear, M. The connectomics of brain disorders. Nat. Rev. Neurosci. 16, 159–172 (2015).
-
Stam, C. J. Modern network science of neurological disorders. Nat. Rev. Neurosci. 15, 683–695 (2014).
-
Achard, S., Salvador, R., Whitcher, B., Suckling, J. & Bullmore, E. A resilient, low-frequency, small-world human brain functional network with highly connected association cortical hubs. J. Neurosci. 26, 63–72 (2006).
-
Watts, D. J. & Strogatz, S. H. Collective dynamics of ‘small-world’ networks. Nature 393, 440–442 (1998).
-
Kaiser, M. & Hilgetag, C. C. Nonoptimal component placement, but short processing paths, due to long-distance projections in neural systems. PLoS Comput. Biol. 2, e95 (2006).
-
Zamora-López, G., Zhou, C. & Kurths, J. Cortical hubs form a module for multisensory integration on top of the hierarchy of cortical networks. Front. Neuroinform. 4, 1 (2010).
-
Papo, D., Zanin, M., Martínez, J. H. & Buldú, J. M. Beware of the small-world neuroscientist! Front. Hum. Neurosci. 10, 96 (2016).
-
Rubinov, M. & Sporns, O. Complex network measures of brain connectivity: uses and interpretations. NeuroImage 52, 1059–1069 (2010).
-
Varshney, L. R., Chen, B. L., Paniagua, E., Hall, D. H. & Chklovskii, D. B. Structural properties of the Caenorhabditis elegans neuronal network. PLoS Comput. Biol. 7, e1001066 (2011).
-
Grifa, A., Baumann, P. S., Thiran, J.-P. & Hagmann, P. Structural connectomics in brain diseases. NeuroImage 80, 515–526 (2013).
-
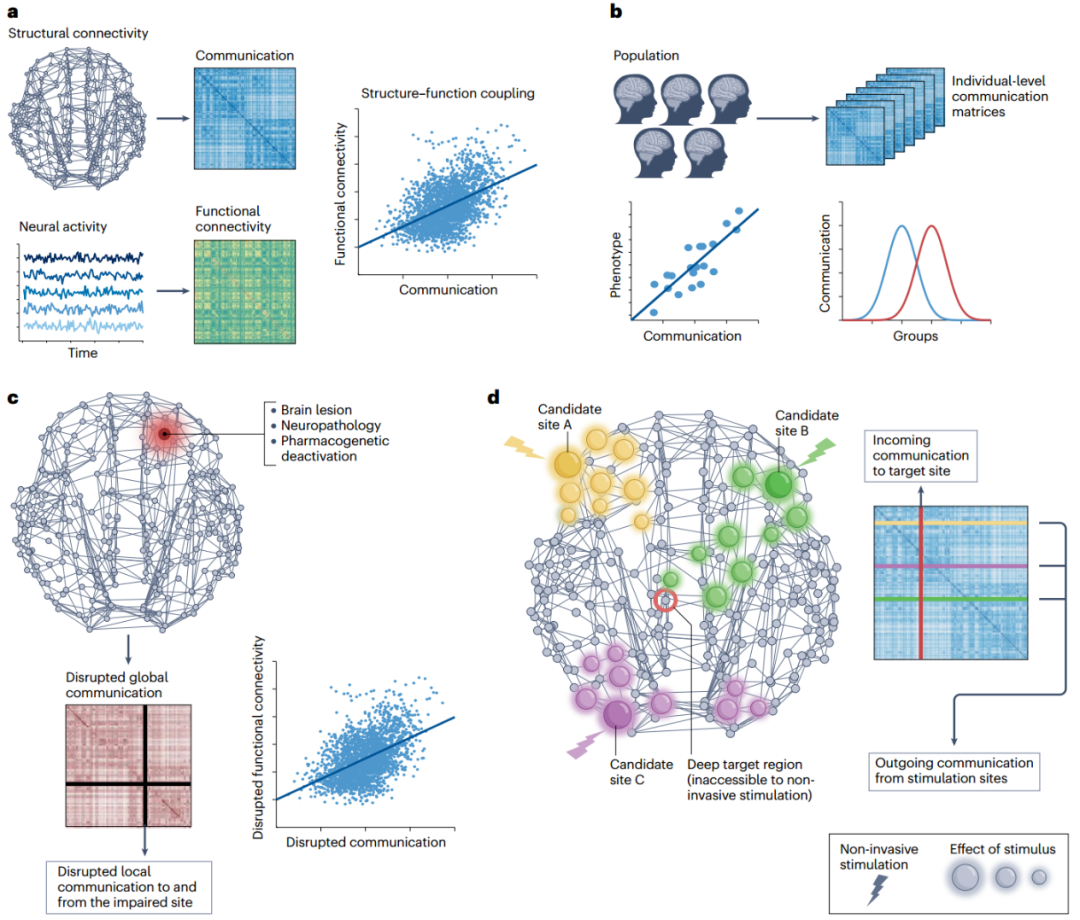
Goñi, J. et al. Resting-brain functional connectivity predicted by analytic measures of network communication. Proc. Natl Acad. Sci. USA 111, 833–838 (2014).
This work was one of the first empirical demonstrations that network communication models can be used to infer functional connectivity from brain network structure.
-
Avena-Koenigsberger, A. et al. Path ensembles and a tradeof between communication eficiency and resilience in the human connectome. Brain Struct. Funct. 222, 603–618 (2016).
-
Seguin, C., van den Heuvel, M. P. & Zalesky, A. Navigation of brain networks. Proc. Natl Acad. Sci. USA 115, 6297–6302 (2018).
This study was the first comprehensive exploration of the network navigation model in neuroscience.
-
Kleinberg, J. M. Navigation in a small world. Nature 406, 845 (2000).
-
Boguna, M., Krioukov, D. & Clafy, K. C. Navigability of complex networks. Nat. Phys. 5, 74–80 (2009).
-
Goñi, J. et al. Exploring the morphospace of communication eficiency in complex networks. PLoS One 8, e58070 (2013).
-
Travers, J. & Milgram, S. An experimental study of the small world problem. Sociometry32, 425–443 (1969).
A prescient and highly influential account of decentralized communication in complex networks.
-
Boguñá, M., Papadopoulos, F. & Krioukov, D. Sustaining the Internet with hyperbolic mapping. Nat. Commun. 1, 62 (2010).
-
Grayson, D. S. et al. The rhesus monkey connectome predicts disrupted functional networks resulting from pharmacogenetic inactivation of the amygdala. Neuron 91, 453–466 (2016).
This paper shows that network communication models can predict global changes in functional connectivity resulting from the deactivation of targeted regions.
-
Betzel, R. F. et al. Structural, geometric and genetic factors predict interregional brain connectivity patterns probed by electrocorticography. Nat. Biomed. Eng. 3, 902–916 (2019).
-
Seguin, C., Tian, Y. & Zalesky, A. Network communication models improve the behavioral and functional predictive utility of the human structural connectome. Netw. Neurosci. 4, 980–1006 (2020).
-
Imms, P. et al. Navigating the link between processing speed and network communication in the human brain. Brain Struct. Funct. 226, 1281–1302 (2021).
-
de Lange, S. C. et al. Shared vulnerability for connectome alterations across psychiatric and neurological brain disorders. Nat. Hum. Behav. 3, 988–998 (2019).
This paper uses a large sample of patients to identify a cross-disorder basis for disrupted brain network communication in neuropsychiatric conditions.
-
Lella, E. & Estrada, E. Communicability distance reveals hidden patterns of Alzheimer’s disease. Netw. Neurosci. 4, 1007–1029 (2020).
-
Wang, X. et al. Synchronization lag in post stroke: relation to motor function and structural connectivity. Netw. Neurosci. 3, 1121–1140 (2019).
-
Hermundstad, A. M. et al. Structural foundations of resting-state and task-based functional connectivity in the human brain. Proc. Natl Acad. Sci. USA 110, 6169–6174 (2013).
-
Cook, S. J. et al. Whole-animal connectomes of both Caenorhabditis elegans sexes. Nature 571, 63–71 (2019).
-
Winding, M. et al. The connectome of an insect brain. Science 379, eadd9330 (2023).
This recent paper was the first to investigate polysynaptic streams of information flow in a complete insect connectome.
-
Alves, P. N. et al. An improved neuroanatomical model of the default-mode network reconciles previous neuroimaging and neuropathological findings. Commun. Biol. 2, 370 (2019).
-
Uddin, L. Q. et al. Residual functional connectivity in the split-brain revealed with resting-state functional MRI. Neuroreport 19, 703–709 (2008).
-
Trebaul, L. et al. Probabilistic functional tractography of the human cortex revisited. Neuroimage 181, 414–429 (2018).
-
Momi, D., Wang, Z. & Grifiths, J. D. TMS-evoked responses are driven by recurrent large-scale network dynamics. eLife 12, e83232 (2023).
-
Laughlin, S. B., de Ruyter van Steveninck, R. R. & Anderson, J. C. The metabolic cost of neural information. Nat. Neurosci. 1, 36–41 (1998).
-
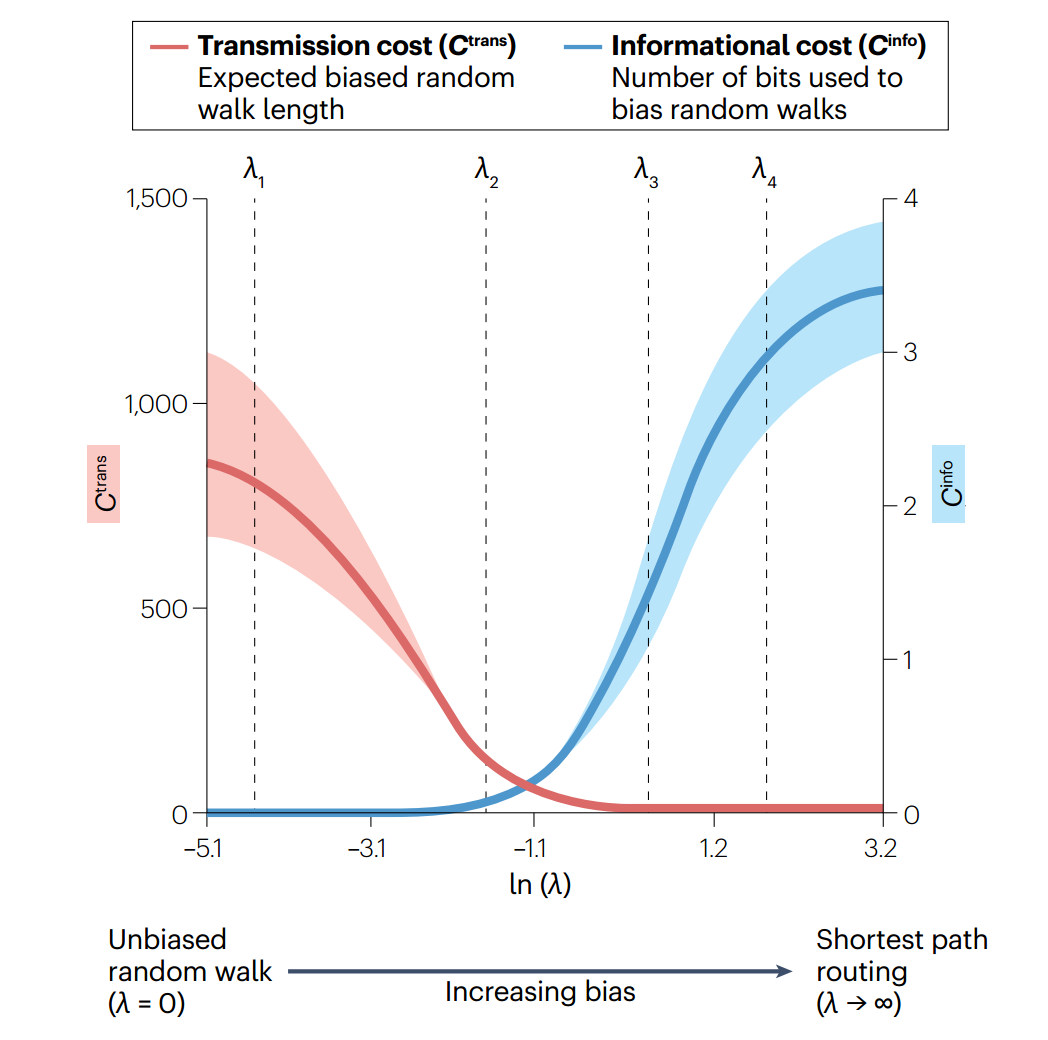
Avena-Koenigsberger, A. et al. A spectrum of routing strategies for brain networks. PLoS Comput. Biol. 15, e1006833 (2019).
This work presents one of the first applications of the biased random walk model in network neuroscience.
-
Avena-Koenigsberger, A. et al. Using Pareto optimality to explore the topology and dynamics of the human connectome. Philos. Trans. R. Soc. Lond. B Biol. Sci. 369, 20130530 (2014).
-
Avena-Koenigsberger, A., Goñi, J., Solé, R. & Sporns, O. Network morphospace. J. R. Soc. Interface 12, 20140881 (2015).
-
Serrano, M. A., Krioukov, D. & Boguñá, M. Self-similarity of complex networks and hidden metric spaces. Phys. Rev. Lett. 100, 078701 (2008).
-
Kleinberg, J. Complex networks and decentralized search algorithms. Proc. Int. Congr. Math. 3, 1019–1044 (2006).
-
Cannistraci, C. V. & Muscoloni, A. Geometrical congruence, greedy navigability and myopic transfer in complex networks and brain connectomes. Nat. Commun. 13, 7308 (2022).
-
Roberts, J. A. et al. The contribution of geometry to the human connectome. NeuroImage124, 379–393 (2016).
-
Stiso, J. & Bassett, D. S. Spatial embedding imposes constraints on neuronal network architectures. Trends Cogn. Sci. 22, 1127–1142 (2018).
-
Vézquez-Rodríguez, B., Liu, Z.-Q., Hagmann, P. & Misic, B. Signal propagation via cortical hierarchies. Netw. Neurosci. 4, 1072–1090 (2020).
-
Boguñá, M. et al. Network geometry. Nat. Rev. Phys. 3, 114–135 (2021).
-
Zheng, M., Allard, A., Hagmann, P., Alemán-Gómez, Y. & Serrano, M. Á. Geometric renormalization unravels self-similarity of the multiscale human connectome. Proc.Natl Acad. Sci. USA 117, 20244–20253 (2020).
-
Allard, A. & Serrano, M. Á. Navigable maps of structural brain networks across species. PLoS Comput. Biol. 16, e1007584 (2020).
-
Duran-Nebreda, S., Johnston, I. G. & Bassel, G. W. Eficient vasculature investment in tissues can be determined without global information. J. R. Soc. Interface 17, 20200137 (2020).
-
Masuda, N., Porter, M. A. & Lambiotte, R. Random walks and difusion on networks. Phys. Rep. 716, 1–58 (2017).
This study is a comprehensive review on random walks dynamics in complex networks.
-
Noh, J. D. & Rieger, H. Random walks on complex networks. Phys. Rev. Lett. 92, 118701 (2004).
-
Estrada, E. & Hatano, N. Communicability in complex networks. Phys. Rev. E Stat. Nonlin. Soft Matter Phys. 77, 036111 (2008)
This seminal work introduced the concept of communicability in complex networks.
-
Estrada, E., Hatano, N. & Benzi, M. The physics of communicability in complex networks. Phys. Rep. 514, 89–119 (2012).
-
Granovetter, M. Threshold models of collective behavior. Am. J. Sociol. 83, 1420–1443 (1978).
-
Nematzadeh, A., Ferrara, E., Flammini, A. & Ahn, Y.-Y. Optimal network modularity for information difusion. Phys. Rev. Lett. 113, 088701 (2014).
-
Mišić, B. et al. Cooperative and competitive spreading dynamics on the human connectome. Neuron 86, 1518–1529 (2015).
-
Worrell, J. C., Rumschlag, J., Betzel, R. F., Sporns, O. & Mišić, B. Optimized connectome architecture for sensory-motor integration. Netw. Neurosci. 1, 415–430 (2018).
-
Shadi, K., Dyer, E. & Dovrolis, C. Multisensory integration in the mouse cortical connectome using a network difusion model. Netw. Neurosci. 4, 1030–1054 (2020).
-
Gómez-Gardeñes, J. & Latora, V. Entropy rate of difusion processes on complex networks. Phys. Rev. E Stat. Nonlin. Soft Matter Phys. 78, 065102 (2008).
-
Lambiotte, R. et al. Flow graphs: interweaving dynamics and structure. Phys. Rev. E Stat. Nonlin. Soft Matter Phys. 84, 017102 (2011).
-
Benigni, B., Gallotti, R. & De Domenico, M. Potential-driven random walks on interconnected systems. Phys. Rev. E 104, 024120 (2021).
-
Yin, C.-Y., Wang, B.-H., Wang, W.-X., Zhou, T. & Yang, H.-J. Eficient routing on scale-free networks based on local information. Phys. Lett. A 351, 220–224 (2006).
-
Csoma, A. et al. Routes obey hierarchy in complex networks. Sci. Rep. 7, 7243 (2017).
-
Deco, G., Jirsa, V. K. & McIntosh, A. R. Emerging concepts for the dynamical organization of resting-state activity in the brain. Nat. Rev. Neurosci. 12, 43–56 (2011).
-
Abdelnour, F., Voss, H. U. & Raj, A. Network difusion accurately models the relationship between structural and functional brain connectivity networks. NeuroImage 90, 335–347 (2014).
-
Cole, M. W., Bassett, D. S., Power, J. D., Braver, T. S. & Petersen, S. E. Intrinsic and task-evoked network architectures of the human brain. Neuron 83, 238–251 (2014).
-
Siddiqi, S. H., Kording, K. P., Parvizi, J. & Fox, M. D. Causal mapping of human brain function. Nat. Rev. Neurosci. 23, 361–375 (2022).
This recent review discusses current eforts and future directions to map causal interactions in the human brain. Concepts outlined in this work may provide avenues for the testing and validation of network communication models.
-
Medaglia, J. D., Lynall, M.-E. & Bassett, D. S. Cognitive network neuroscience. J. Cogn. Neurosci. 27, 1471–1491 (2015).
-
Smith, S. M. et al. A positive–negative mode of population covariation links brain connectivity, demographics and behavior. Nat. Neurosci. 18, 1565–1567 (2015).
-
Fornito, A., Zalesky, A. & Breakspear, M. Graph analysis of the human connectome: promise, progress, and pitfalls. NeuroImage 80, 426–444 (2013).
-
Popp, J. L. et al. Structural–functional brain network coupling predicts human cognitive ability. Preprint at bioRxiv https://doi.org/10.1101/2023.02.09.527639 (2023).
-
Zorlu, N. et al. Abnormal structural network communication reflects cognitive deficits in schizophrenia. Brain Topogr. 36, 294–304 (2023).
-
Imms, P. et al. The structural connectome in traumatic brain injury: a meta-analysis of graph metrics. Neurosci. Biobehav. Rev. 99, 128–137 (2019)
-
Imms, P. et al. Exploring personalized structural connectomics for moderate to severe traumatic brain injury. Netw. Neurosci. 7, 160–183 (2023).
-
Kuceyeski, A. et al. The application of a mathematical model linking structural and functional connectomes in severe brain injury. NeuroImage Clin. 11, 635–647 (2016).
-
Kuceyeski, A. F., Jamison, K. W., Owen, J. P., Raj, A. & Mukherjee, P. Longitudinal increases in structural connectome segregation and functional connectome integration are associated with better recovery after mild TBI. Hum. Brain Mapp. 40, 4441–4456 (2019).
-
Albert, R., Jeong, H. & Barabasi, A. L. Error and attack tolerance of complex networks. Nature 406, 378–382 (2000).
-
Palop, J. J. & Mucke, L. Amyloid-beta-induced neuronal dysfunction in Alzheimer’s disease: from synapses toward neural networks. Nat. Neurosci. 13, 812–818 (2010).
-
Raj, A., Kuceyeski, A. & Weiner, M. A network difusion model of disease progression in dementia. Neuron 73, 1204–1215 (2012).
-
Meier, J. M. et al. Connectome-based propagation model in amyotrophic lateral sclerosis. Ann. Neurol. 87, 725–738 (2020).
-
Vogel, J. W. et al. Spread of pathological tau proteins through communicating neurons in human Alzheimer’s disease. Nat. Commun. 11, 2612 (2020).
-
Zheng, Y.-Q. et al. Local vulnerability and global connectivity jointly shape neurodegenerative disease propagation. PLoS Biol. 17, e3000495 (2019).
-
Wannan, C. M. J. et al. Evidence for network-based cortical thickness reductions in schizophrenia. Am. J. Psychiatry 176, 552–563 (2019).
-
Shafiei, G. et al. Spatial patterning of tissue volume loss in schizophrenia reflects brain network architecture. Biol. Psychiatry 87, 727–735 (2020).
-
Miller, K. L. et al. Multimodal population brain imaging in the UK Biobank prospective epidemiological study. Nat. Neurosci. 19, 1523–1536 (2016).
-
Sina Mansour, L., Di Biase, M. A., Smith, R. E., Zalesky, A. & Seguin, C. Connectomes for 40,000 UK Biobank participants: a multi-modal, multi-scale brain network resource. Preprint at bioRxiv https://doi.org/10.1101/2023.03.10.532036 (2023).
-
Keller, C. J. et al. Mapping human brain networks with cortico-cortical evoked potentials. Philos. Trans. R. Soc. Lond. B Biol. Sci. 369, 20130528 (2014).
-
Sydnor, V. J. et al. Cortical–subcortical structural connections support transcranial magnetic stimulation engagement of the amygdala. Sci. Adv. 8, eabn5803 (2022).
-
Ozdemir, R. A. et al. Individualized perturbation of the human connectome reveals reproducible biomarkers of network dynamics relevant to cognition. Proc. Natl Acad. Sci. USA 117, 8115–8125 (2020).
-
Veit, M. J. et al. Temporal order of signal propagation within and across intrinsic brain networks. Proc. Natl Acad. Sci. USA 118, e2105031118 (2021).
-
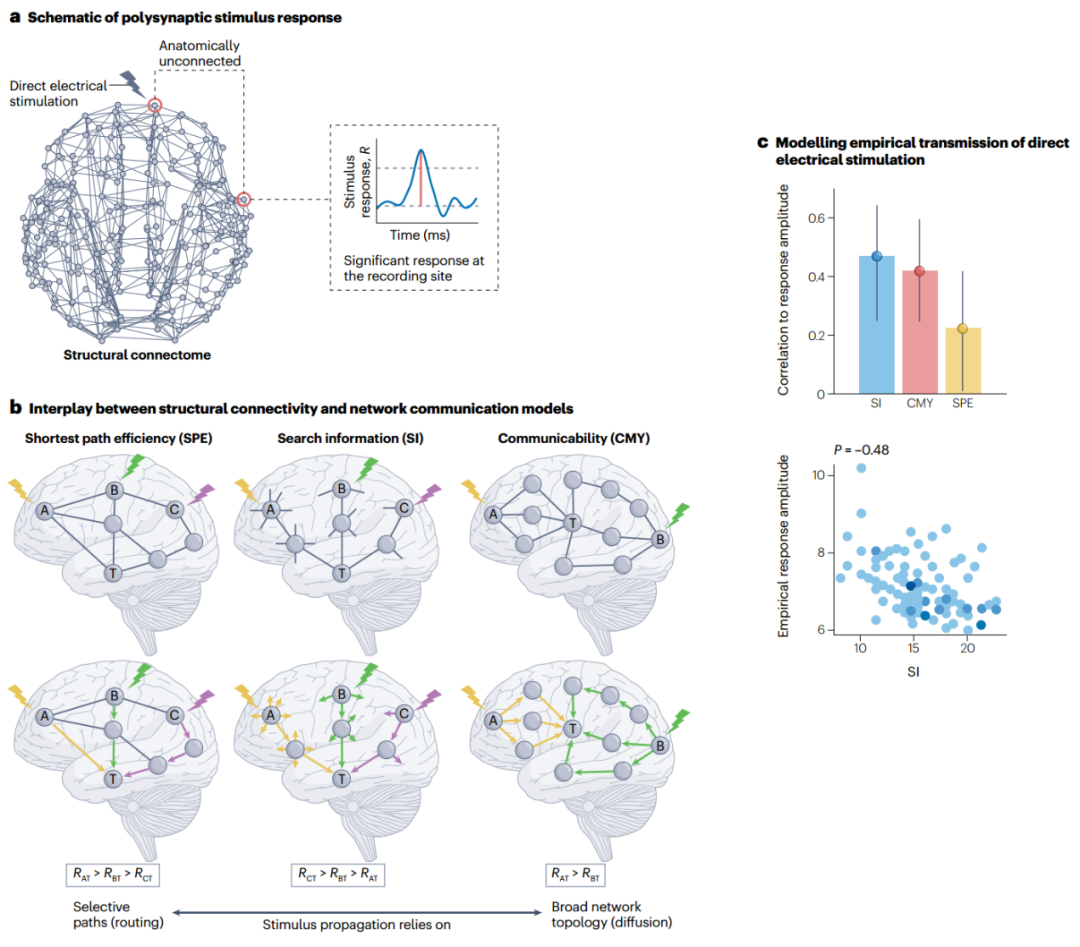
Seguin, C. et al. Communication dynamics in the human connectome shape the cortex-wide propagation of direct electrical stimulation. Neuron 111, 1391–1401.e5 (2023).
The first paper to show that network communication models can predict the propagation of direct electrical stimulation through the human brain.
-
Siddiqi, S. H., Taylor, J. J., Horn, A. & Fox, M. D. Bringing human brain connectomics to clinical practice in psychiatry. Biol. Psychiatry 93, 386–387 (2023).
-
Horn, A. & Fox, M. D. Opportunities of connectomic neuromodulation. NeuroImage 221, 117180 (2020).
-
Horn, A. et al. Connectivity predicts deep brain stimulation outcome in Parkinson disease. Ann. Neurol. 82, 67–78 (2017).
-
Cash, R. F. H. et al. Subgenual functional connectivity predicts antidepressant treatment response to transcranial magnetic stimulation: independent validation and evaluation of personalization. Biol. Psychiatry 86, e5–e7 (2019).
-
Fox, M. D., Buckner, R. L., White, M. P., Greicius, M. D. & Pascual-Leone, A. Eficacy of transcranial magnetic stimulation targets for depression is related to intrinsic functional connectivity with the subgenual cingulate. Biol. Psychiatry 72, 595–603 (2012).
-
Schelling, T. C. Dynamic models of segregation. J. Math. Sociol. 1, 143–186 (1971).
-
Helbing, D., Farkas, I. & Vicsek, T. Simulating dynamical features of escape panic. Nature407, 487–490 (2000).
-
Zamani Esfahlani, F., Faskowitz, J., Slack, J., Mišić, B. & Betzel, R. F. Local structure–function relationships in human brain networks across the lifespan. Nat. Commun. 13, 2053 (2022).
This paper carries out a comprehensive comparison of the extent to which diferent network communication models can explain patterns of functional connectivity.
-
Mišic, B. et al. Network-based asymmetry of the human auditory system. Cereb. Cortex28, 2655–2664 (2018).
-
Seguin, C., Mansour, C. S., Sporns, O., Zalesky, A. & Calamante, F. Network communication models narrow the gap between the modular organization of structural and functional brain networks. NeuroImage 257, 119323 (2022).
-
Amico, E. et al. Toward an information theoretical description of communication in brain networks. Netw. Neurosci. 5, 646–665 (2021).
-
Hao, Y. & Graham, D. Creative destruction: sparse activity emerges on the mammal connectome under a simulated communication strategy with collisions and redundancy. Netw. Neurosci. 4, 1055–1071 (2020).
-
Hillary, F. G. & Grafman, J. H. Injured brains and adaptive networks: the benefits and costs of hyperconnectivity. Trends Cogn. Sci. 21, 385–401 (2017).
-
Park, B.-Y. et al. Signal difusion along connectome gradients and inter-hub routing diferentially contribute to dynamic human brain function. NeuroImage 224, 117429 (2021).
-
Bazinet, V., Vos de Wael, R., Hagmann, P., Bernhardt, B. C. & Misic, B. Multiscale communication in cortico-cortical networks. NeuroImage 243, 118546 (2021).
-
Betzel, R. F., Faskowitz, J., Mišić, B., Sporns, O. & Seguin, C. Multi-policy models of interregional communication in the human connectome. Preprint at bioRxiv https://doi.org/10.1101/2022.05.08.490752 (2022).
-
Mišić, B., Sporns, O. & McIntosh, A. R. Communication eficiency and congestion of signal trafic in large-scale brain networks. PLoS Comput. Biol. 10, e1003427 (2014).
-
Grifa, A. et al. Transient networks of spatio-temporal connectivity map communication pathways in brain functional systems. NeuroImage 155, 490–502 (2017).
This work uses an innovative multilayer framework to track the time-resolved propagation of functional activity and relates it to communication via structural connectivity.
-
Liu, Z.-Q. et al. Time-resolved structure–function coupling in brain networks. Commun. Biol. 5, 532 (2022).
-
de Lange, S. C., de Reus, M. A. & van den Heuvel, M. P. The Laplacian spectrum of neural networks. Front. Comput. Neurosci. 7, 189 (2014).
-
Becker, C. O. et al. Spectral mapping of brain functional connectivity from difusion imaging. Sci. Rep. 8, 1411 (2018).
-
Abdelnour, F., Dayan, M., Devinsky, O., Thesen, T. & Raj, A. Functional brain connectivity is predictable from anatomic network’s Laplacian eigen-structure. NeuroImage 172, 728–739 (2018).
-
Deslauriers-Gauthier, S., Zucchelli, M., Frigo, M. & Deriche, R. A unified framework for multimodal structure–function mapping based on eigenmodes. Med. Image Anal. 66, 101799 (2020).
-
Tewarie, P. et al. Mapping functional brain networks from the structural connectome: relating the series expansion and eigenmode approaches. NeuroImage 216, 116805 (2020).
-
Meier, J. et al. The epidemic spreading model and the direction of information flow in brain networks. NeuroImage 152, 639–646 (2017).
-
Benigni, B., Ghavasieh, A., Corso, A., d’Andrea, V. & De Domenico, M. Persistence of information flow: a multiscale characterization of human brain. Netw. Neurosci. 5, 831–850 (2021).
-
Graham, D. & Rockmore, D. The packet switching brain. J. Cogn. Neurosci. 23, 267–276 (2011).
-
Betzel, R. F., Gu, S., Medaglia, J. D., Pasqualetti, F. & Bassett, D. S. Optimally controlling the human connectome: the role of network topology. Sci. Rep. 6, 30770 (2016).
-
Deco, G. et al. Resting-state functional connectivity emerges from structurally and dynamically shaped slow linear fluctuations. J. Neurosci. 33, 11239–11252 (2013).
-
Honey, C. J. et al. Predicting human resting-state functional connectivity from structural connectivity. Proc. Natl Acad. Sci. USA 106, 2035–2040 (2009).
-
Muldoon, S. F. et al. Stimulation-based control of dynamic brain networks. PLoS Comput. Biol. 12, e1005076 (2016).
-
Gollo, L. L., Roberts, J. A. & Cocchi, L. Mapping how local perturbations influence systems-level brain dynamics. NeuroImage 160, 97–112 (2017).
-
Stiso, J. et al. White matter network architecture guides direct electrical stimulation through optimal state transitions. 2018 Conference on Cognitive Computational Neuroscience. https://doi.org/10.32470/ccn.2018.1028-0 (2018).
-
Aerts, H., Fias, W., Caeyenberghs, K. & Marinazzo, D. Brain networks under attack: robustness properties and the impact of lesions. Brain 139, 3063–3083 (2016).
-
Srivastava, P. et al. Models of communication and control for brain networks: distinctions, convergence, and future outlook. Netw. Neurosci. 4, 1122–1159 (2020).
A recent review highlighting opportunities for synergy between diferent computational avenues to model brain function, including network communication, dynamical systems and control theory.
-
Finger, H., Gast, R., Gerlof, C., Engel, A. K. & König, P. Probing neural networks for dynamic switches of communication pathways. PLOS Comput. Biol. 15, e1007551 (2019).
-
Pope, M., Seguin, C., Varley, T. F., Faskowitz, J. & Sporns, O. Co-evolving dynamics and topology in a coupled oscillator model of resting brain function. Preprint at bioRxivhttps://doi.org/10.1101/2023.01.31.526514 (2023).
-
Osmanlıoğlu, Y. et al. System-level matching of structural and functional connectomes in the human brain. Neuroimage 199, 93–104 (2019).
-
Seguin, C., Razi, A. & Zalesky, A. Inferring neural signalling directionality from undirected structural connectomes. Nat. Commun. 10, 4289 (2019).
-
Vázquez-Rodríguez, B. et al. Gradients of structure–function tethering across neocortex. Proc. Natl Acad. Sci. USA 116, 21219–21227 (2019).
-
Tewarie, P. et al. Predicting time‐resolved electrophysiological brain networks from structural eigenmodes. Hum. Brain Mapp. 43, 4475–4491 (2022).
-
Nakuci, J., McGuire, M., Schweser, F., Poulsen, D. & Muldoon, S. F. Diferential patterns of change in brain connectivity resulting from severe traumatic brain injury. Brain Connect.12, 799–811 (2022).
-
Li, Y. et al. Difusion tensor imaging based network analysis detects alterations of neuroconnectivity in patients with clinically early relapsing-remitting multiple sclerosis. Hum. Brain Mapp. 34, 3376–3391 (2013).
-
Crofts, J. J. et al. Network analysis detects changes in the contralesional hemisphere following stroke. NeuroImage 54, 161–169 (2011).
-
Andreotti, J. et al. Validation of network communicability metrics for the analysis of brain structural networks. PLoS One 9, e115503 (2014).
-
Latora, V. & Marchiori, M. Eficient behavior of small-world networks. Phys. Rev. Lett. 87, 198701 (2001).
-
Muscoloni, A., Thomas, J. M., Ciucci, S., Bianconi, G. & Cannistraci, C. V. Machine learning meets complex networks via coalescent embedding in the hyperbolic space. Nat. Commun. 8, 1615 (2017).
-
Rosvall, M., Grönlund, A., Minnhagen, P. & Sneppen, K. Searchability of networks. Phys. Rev. E Stat. Nonlin. Soft Matter Phys. 72, 046117 (2005).
This paper first proposed the concept of search information and applied it to complex networks.
-
Sneppen, K., Trusina, A. & Rosvall, M. Hide-and-seek on complex networks. Europhys. Lett. 69, 853–859 (2005).
-
Kaiser, M., Görner, M. & Hilgetag, C. C. Criticality of spreading dynamics in hierarchical cluster networks without inhibition. N. J. Phys. 9, 110 (2007).
-
Pei, S. & Makse, H. A. Spreading dynamics in complex networks. J. Stat. Mech. Theory Exp. 2013, P12002 (2013).
-
Wook Yoo, S. et al. A network flow-based analysis of cognitive reserve in normal ageing and Alzheimer’s disease. Sci. Rep. 5, 10057 (2015).
-
Kaiser, M., Martin, R., Andras, P. & Young, M. P. Simulation of robustness against lesions of cortical networks. Eur. J. Neurosci. 25, 3185–3192 (2007).