PNAS 速递:蛋白质结构预测中 transformer 的变换能力
关键词:Transformer神经网络,蛋白质结构预测,结构生物学


论文题目:The transformative power of transformers in protein structure prediction 期刊名称:PNAS
斑图地址:https://pattern.swarma.org/paper/4959892e-2ffe-11ee-a742-0242ac17000d
论文地址:https://www.pnas.org/doi/10.1073/pnas.2303499120
Transformer神经网络以前所未有的高准确性预测蛋白质结构,彻底改变了结构生物学。这方面的标志性事件是2020年,DeepMind的AlphaFold2方法在从氨基酸序列预测蛋白质的三维结构方面取得了重大科学突破。AlphaFold2框架的核心是由注意力机制驱动的Transformer神经网络组成。Transformer架构之所以非常强大,是因为它能够模拟输入序列中超出其顺序邻域的长程关系。在AlphaFold成功的同时,RoseTTAFold、OmegaFold等框架也在快速发展。自AlphaFold2在2020年取得突破以来,学界在预测蛋白质结构方面取得了多大的进展?尤其是Transformer架构在其中起到多大作用?这些成为了新的问题。
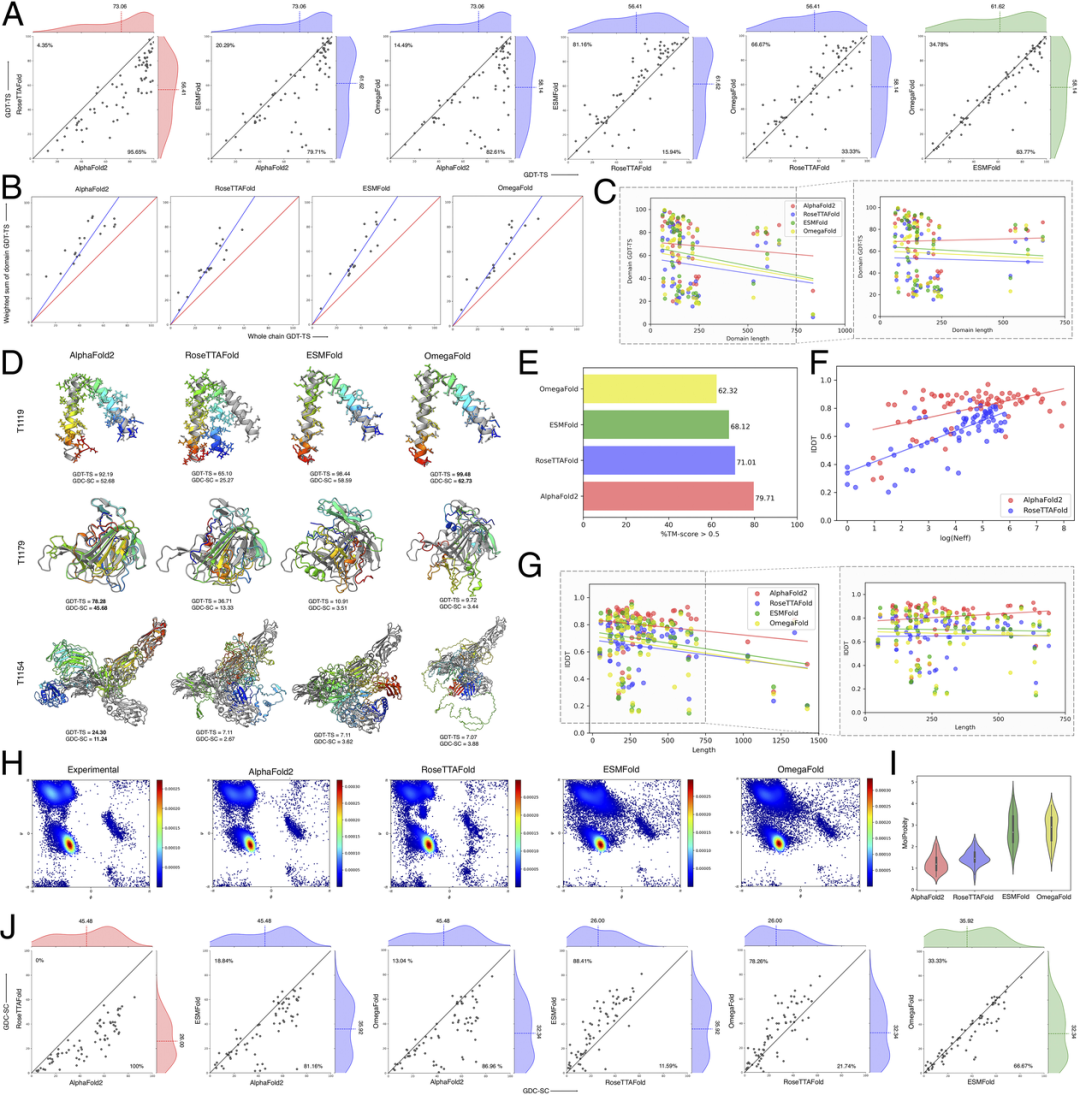
在结构预测界,广泛认可需要进行盲目结构预测测试,以客观评估新的蛋白质结构预测方法的性能。最近结束的CASP15会议为评估这些新兴方法提供了一个优秀的测试平台。新发表于PNAS的一篇研究,对69个CASP15单链蛋白质目标的预测建模性能进行了基准测试。首先,研究者下载了所有方法的开源软件实现,并使用默认参数设置的建模流程进行了完全自动化的模式,没有任何手动干预。其次,研究者通过直接将全长预测与实验坐标进行比较,而不是将其分割成域,来评估单链预测建模的准确性。对多域蛋白质进行了额外的领域级别分析,以检查各个领域及其结构的相对准确性。第三,研究者使用的是在CASP15之前发布的静态数据库、库和模型权重,没有中间更新。
该论文展示了各种方法的主干准确性以及它们之间使用全局距离测试(GDT-TS)指标的对比结果,如图1所示。其中AlphaFold2以最高的平均GDT-TS得分73.06取得了最佳表现,明显优于其他所有方法。ESMFold在主干定位方面表现出第二好的性能,平均GDT-TS得分为61.62。有趣的是,基于PLM(protein language models,蛋白质语言模型)的ESMFold在超过80%的情况下优于基于MSA(multiple sequence alignment,多重序列比对)的RoseTTAFold,并获得了更高的平均GDT-TS得分。相比之下,AlphaFold2在近80%的情况下优于ESMFold。在这两种基于PLM的方法中,ESMFold在主干定位方面的整体准确性优于OmegaFold。
尽管AlphaFold2以基于Transformer的神经网络在蛋白质结构预测方面取得了显著进展,但该研究揭示了两个持续存在的问题。首先,对于具有复杂拓扑结构的大型多域蛋白质的准确预测仍然具有挑战性。对于其中一些目标,没有一种方法能够准确预测出适当的域定向和整体拓扑结构。通过模拟蛋白质间相互作用的原则来调整Transformer模型的架构,可能对于大型多域蛋白质是必要的。其次,目前所有方法在侧链定位的准确性上仍然较低。结构预测算法将受益于准确的侧链排列,这可能要通过在神经架构中加入侧链优化来实现。
作者提出,结合MSA(multiple sequence alignment,多重序列比对)和PLM(protein language models,蛋白质语言模型)两者的优势,可能是进一步推进结构生物学中蛋白质预测模型的途径。
大模型与生物医学:
AI + Science第二季读书会启动
详情请见:
推荐阅读