Nat. Commun. 速递:面向空间组学的两阶段对齐与拼接方法
董弘禹 | 作者

论文题目:SANTO: a coarse-to-fine alignment and stitching method for spatial omics
论文来源:Nature Communications
论文链接:https://www.nature.com/articles/s41467-024-50308-x
随着空间组学技术的蓬勃发展,对平面2D切片进行对齐与拼接,进而形成3D空间模型以发现全局特征,已成为空间组学分析中不可或缺的步骤。然而,现有的方法在处理大规模空间组学数据集时面临挑战,尤其是基于荧光与图像技术产生的数据,在分析时往往耗时过多,分析结果也不尽准确。
鉴于此,来自阿卜杜拉国王科技大学和耶鲁大学的研究人员提出了一种针对空间组学的从粗到精的两阶段对齐策略——SANTO。SANTO分为两个阶段(图1),首先在粗调阶段,通过计算相关系数快速匹配识别两切片重叠区域;随后在精调阶段,通过使用动态图卷积神经网络(DGCNN) 整合空间特征与基因表达特征,进一步对两切片进行精细化对齐。
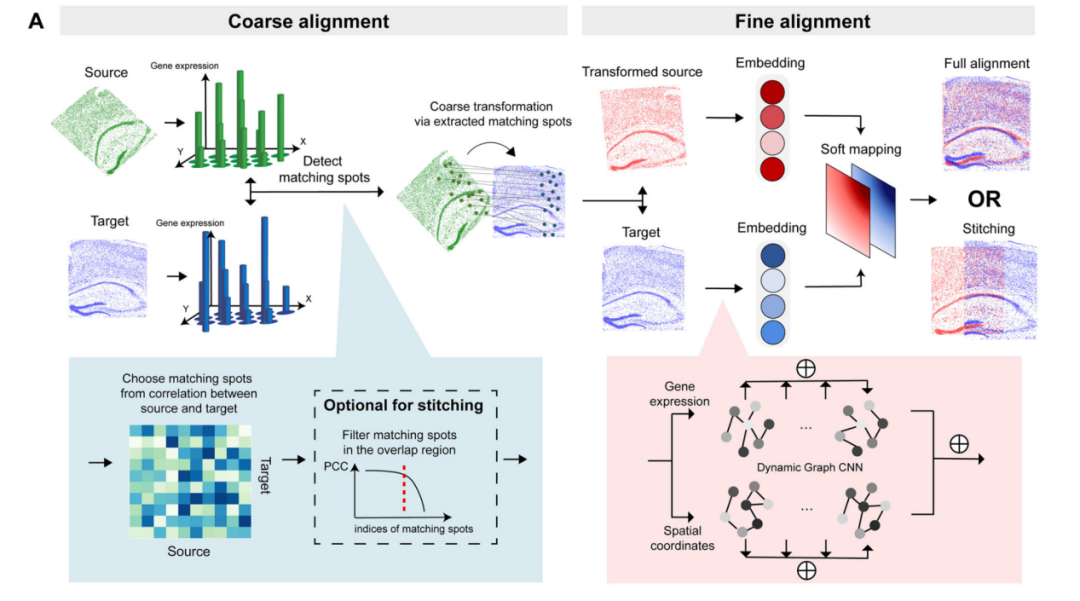
图1 SANTO的算法示意图
研究者们通过广泛实验验证了SANTO相比现有方法的卓越性能。首先,他们在各种数据集上同已发表的各种方法实施了基准测评(图2),测评结果展示SANTO在对齐任务与拼接任务的精确度和鲁棒性都超过了现有方法。
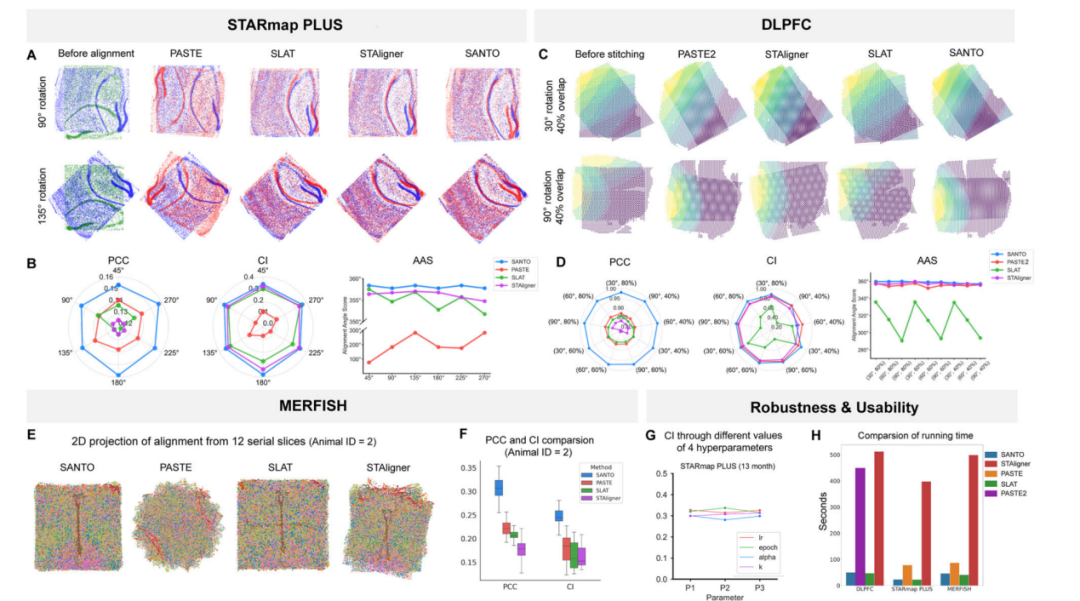
图2 SANTO与其他现有方法的基准测评比较
验证了SANTO模型的高性能后,研究人员使用此工具开展了丰富的下游应用。具体而言,他们对来自同一组织(乳腺癌样本)的跨平台的切片(10x Xenium和Visium)进行了对齐和拼接(图3),确定了更多样化的细胞类型组成,并通过空间细胞通讯分析,更精细探索了肿瘤微环境,为临床治疗提供新的指导。
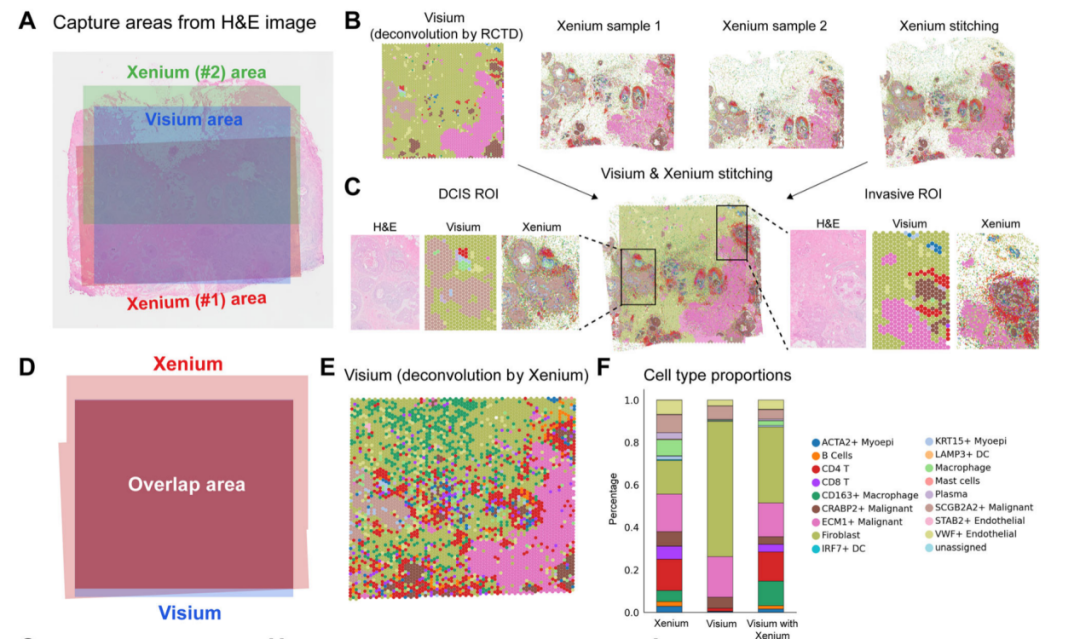
图3 SANTO成功拼接乳腺癌样本的跨平台切片,探索肿瘤微环境
研究人员进一步将模型应用于对齐三维时空组学数据,进而揭示组织发育进程。通过对齐两个小鼠胚胎样本(三维样本,分别为E14.5和E15.5时期,每个样本由连续4片二维切片构成),模型能够揭示肝、肺、心脏等器官的时空演化路径,有助于衰老和癌症进化的研究(图4)。
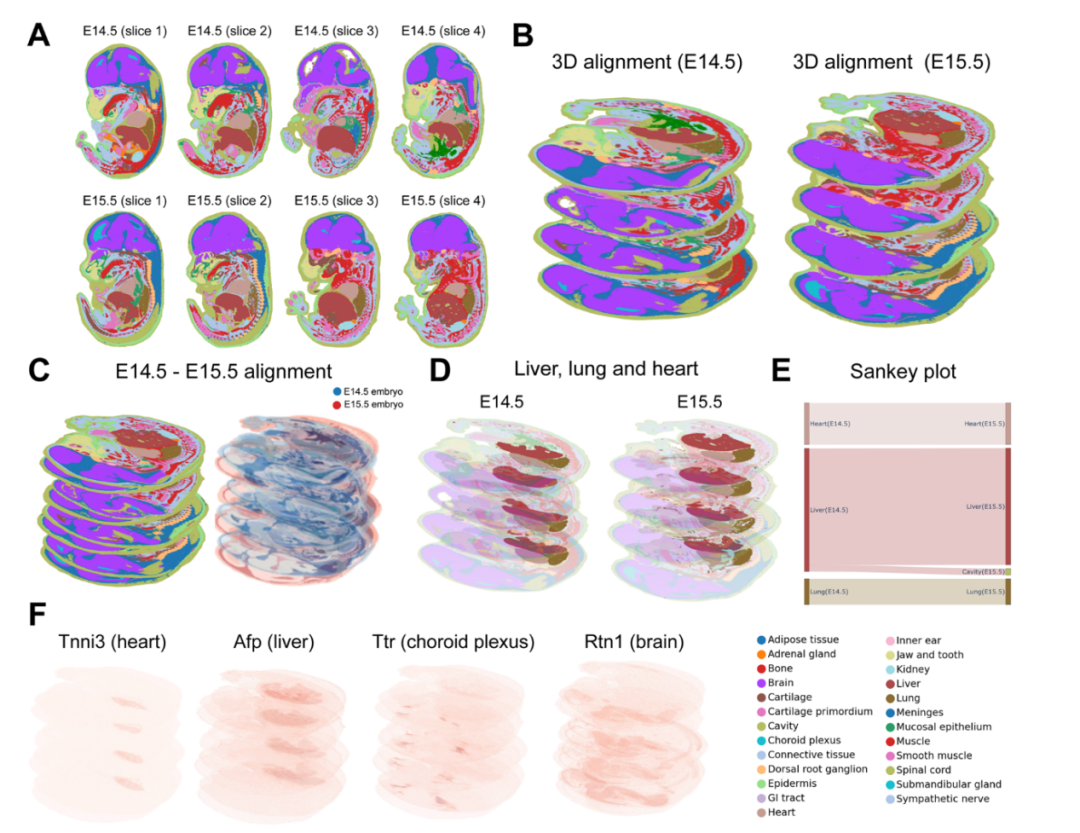
图4 利用SANTO实现3D-to-3D的时空对齐
最后,研究者们使用SANTO将基因表达和组蛋白测序两种模态的切片进行对齐,实现了跨模态整合,展现了SANTO能够进行多样广泛的对齐任务(图5)。
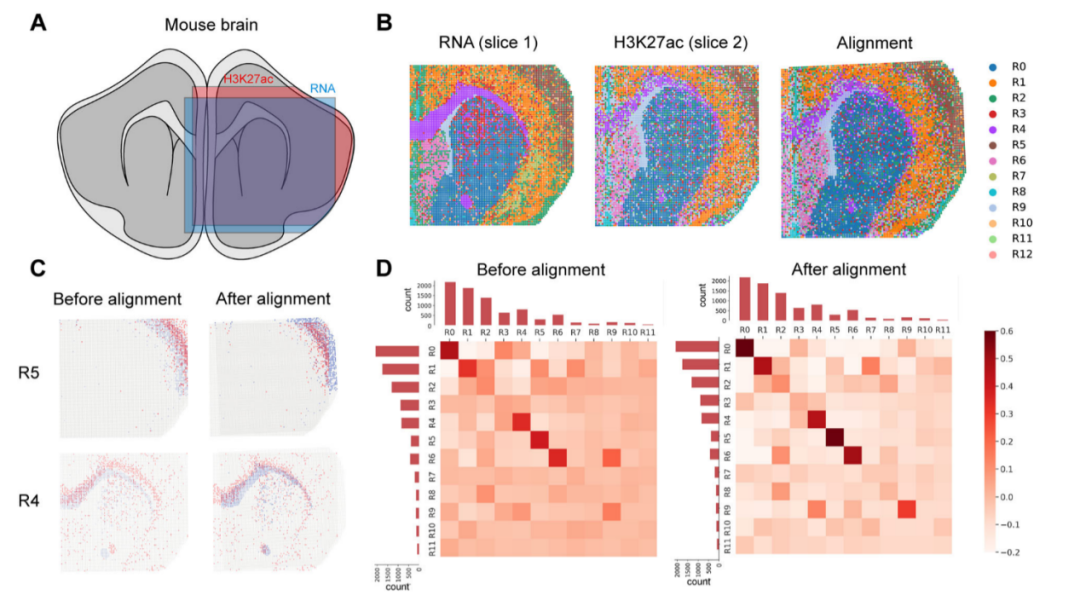
图5 利用SANTO实现跨模态对齐
总之,在本研究中,研究人员研发了一个通用框架——SANTO,来解决空间组学的对齐和拼接问题。全面的基准测试揭示了模型在准确性,稳健性和可用性方面的卓越性能,通过跨平台拼接、3D-to-3D时空对齐和跨模态对齐等任务,进一步展示了SANTO在各种具有挑战性的应用中的强大功能。
生命复杂性读书会招募中
集智俱乐部联合西湖大学理学院及交叉科学中心讲席教授汤雷翰,国家蛋白质科学中心(北京)副研究员常乘、李杨,香港浸会大学助理教授唐乾元,北京大学前沿交叉学科研究院研究员林一瀚,中国科学院分子细胞科学卓越创新中心博士后唐诗婕,共同发起「生命复杂性:生命复杂系统的构成原理」读书会,从微观细胞尺度、介观组织器官尺度,到宏观人体尺度,梳理生命科学领域中的重要问题以及重要数据,希望促进统计物理、机器学习方法研究者和组学研究者之间的深度交流,建立跨学科合作关系,激发新的研究思路和合作项目。读书会从2024年8月6日开始,每周二晚19:00-21:00进行,持续时间预计10-12周。欢迎对这个生命科学、物理学、复杂科学、计算机科学深度交叉的前沿领域感兴趣的朋友加入!

点击“阅读原文”,报名读书会










