关键词:空间转录组学,跨物种整合,全脑图谱,异质图网络,对比学习
![]()
论文题目:Whole brain alignment of spatial transcriptomics between humans and mice
论文来源:Nature Communications
论文链接:https://doi.org/10.1038/s41467-024-50608-2
在生物学实验中,由于伦理限制,科学家们经常使用模式动物替代人体进行实验。在用于模拟人类大脑的各物种中,小鼠由于其生命周期短、和人相似程度高而应用广泛。然而,由于8000万年前开始的重大进化分歧[1],小鼠和人的生命状态仍有很大差别,此外,在众多历史研究中,科学家们对小鼠和人的解剖术语不尽相同,这都给未来的研究带来了很多不便之处。因此,亟需将小鼠和人类的大脑结构特征进行跨物种对齐。之前的方法通过单细胞转录组(scRNA-seq)进行了跨物种对齐[2],然而并未引入空间信息,此外,许多研究都局限于具体的组织层面,目前少有针对于全脑空间图谱进行对齐的工作。
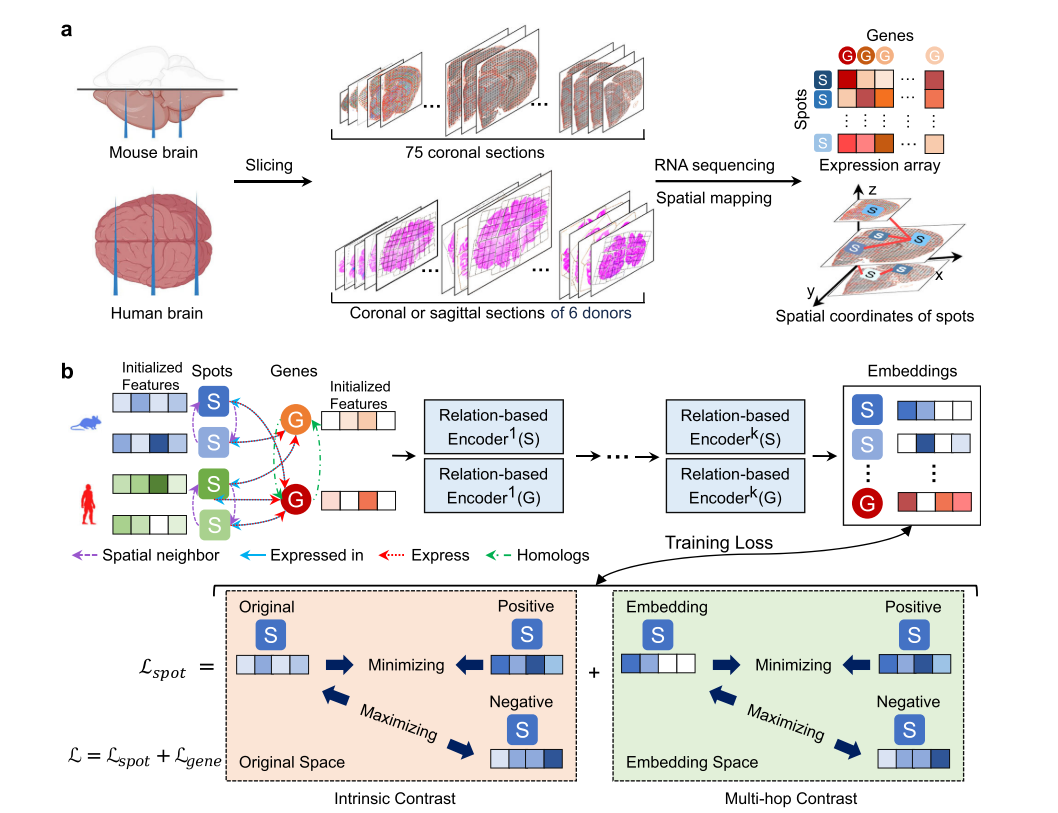
鉴于此,来自中国科学院数学与系统科学研究院张世华团队与复旦大学张淑芹团队合作开发了BrainAlign,一种跨物种比对全脑空间图谱的方法(图1)。BrainAlign采用基于测序的(Microarray sequencing based)人和鼠数据集进行计算。模型分为以下几个部分:
2. 构建以测序点(Spot,即下图的S)和基因(Gene,即下图的G)为节点,各种关系为边(测序点空间彼此邻近则有边相连,基因在测序点中表达则有边相连,基因之间为同源关系则有边相连)的异构图,通过图注意力机制进行消息聚合;
3. 利用获得的隐空间嵌入(latent embedding)和随机选取的正负样本对构建对比学习损失函数,使得正样本向量相似度增加,负样本相似度降低,从而优化隐空间嵌入;
4. 为了能够获取到更广泛的空间特征,模型还考虑了多跳关系(multi-hop relation),即考虑“邻居的邻居”对整个图进行更泛化的消息聚合,进一步增加了特征提取能力。
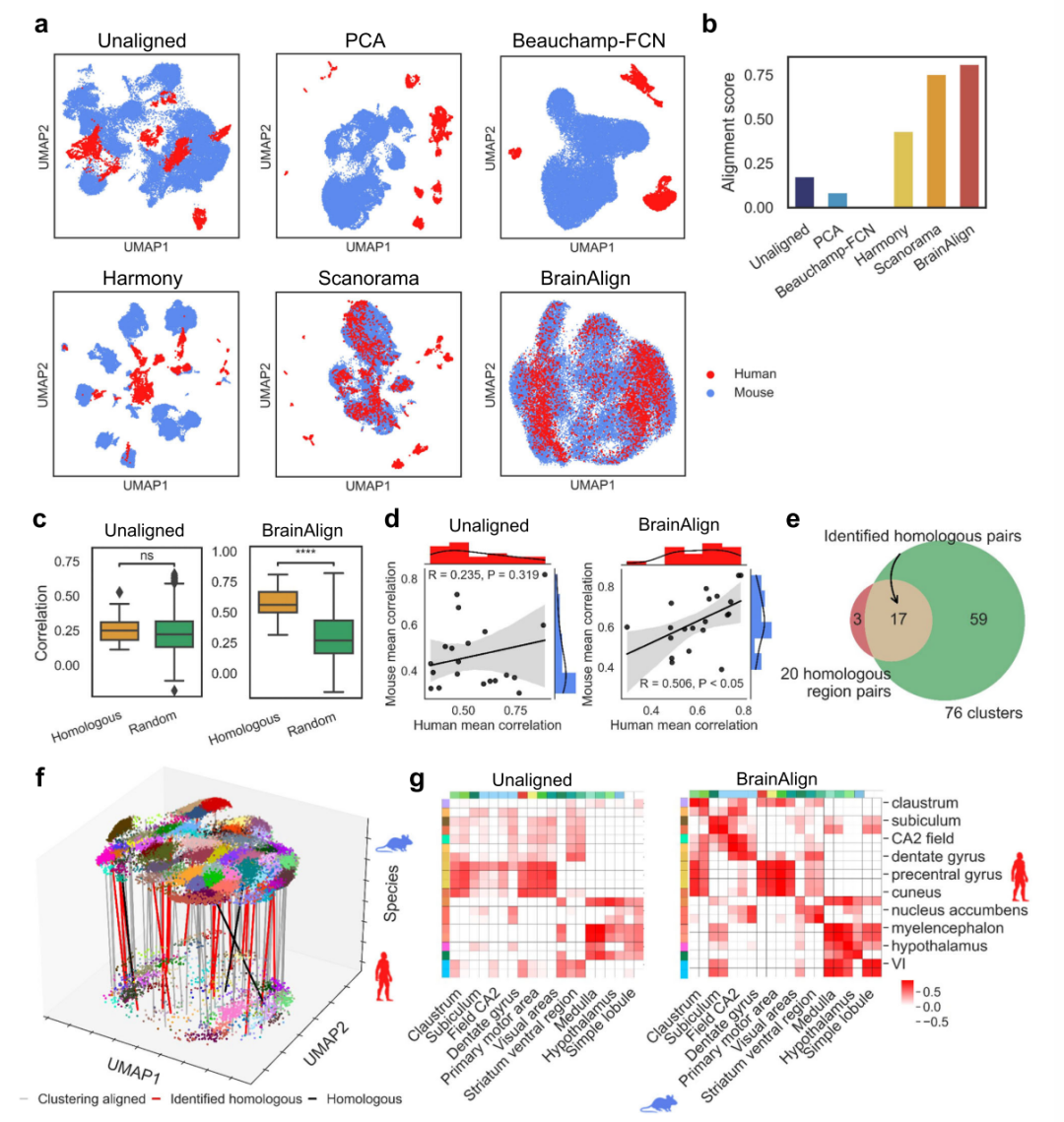
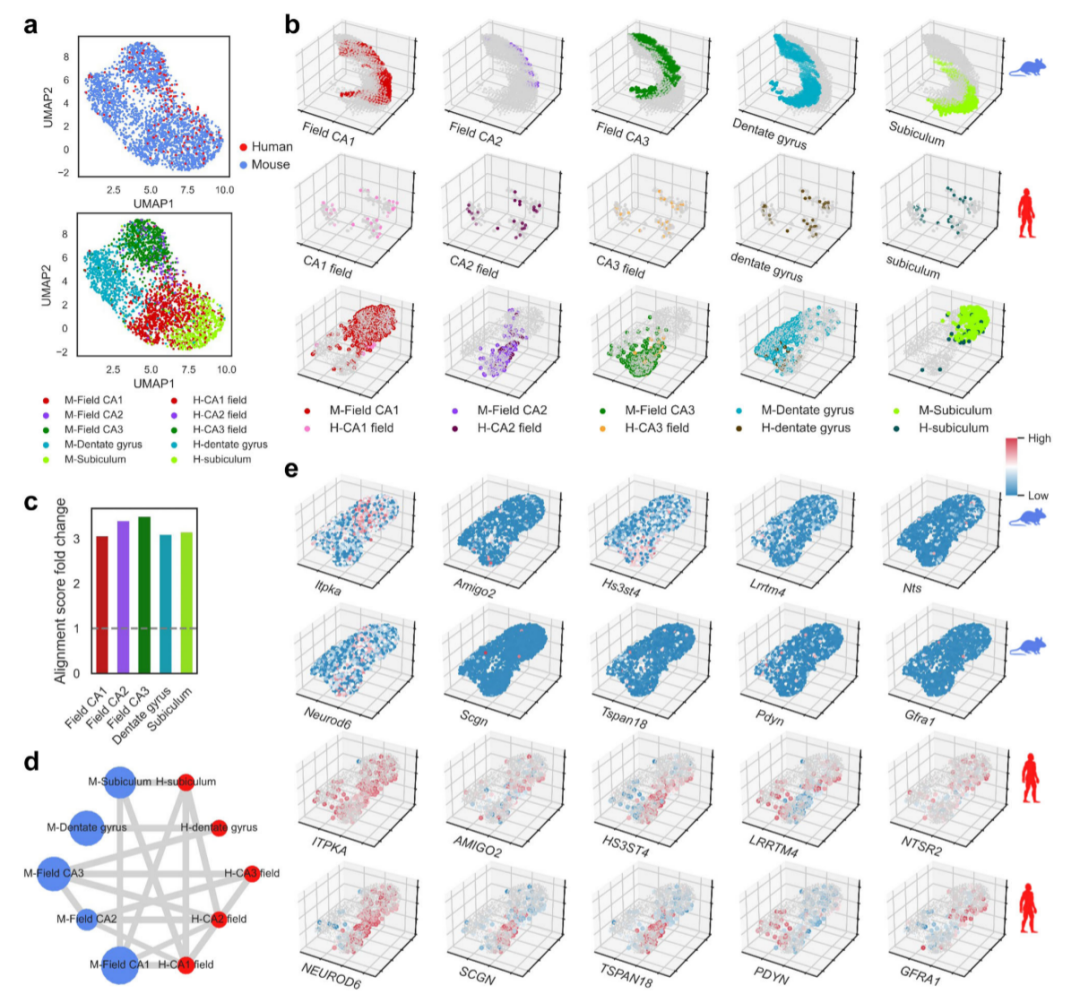
研究者们首先通过将算法运行后得到的隐空间嵌入投影到二维UMAP图上,并和传统的批次效应校正算法(Harmony等)进行比较,发现BrainAlign的效果较好,其他算法不能捕捉到脑区高度异质性的结构,因此效果不佳(图2a)。通过计算匹配分数和同源脑区相关性,作者们发现BrainAlign能够恰当地配对同源脑区,相比于初始情况有很大提升(图2b – g)。
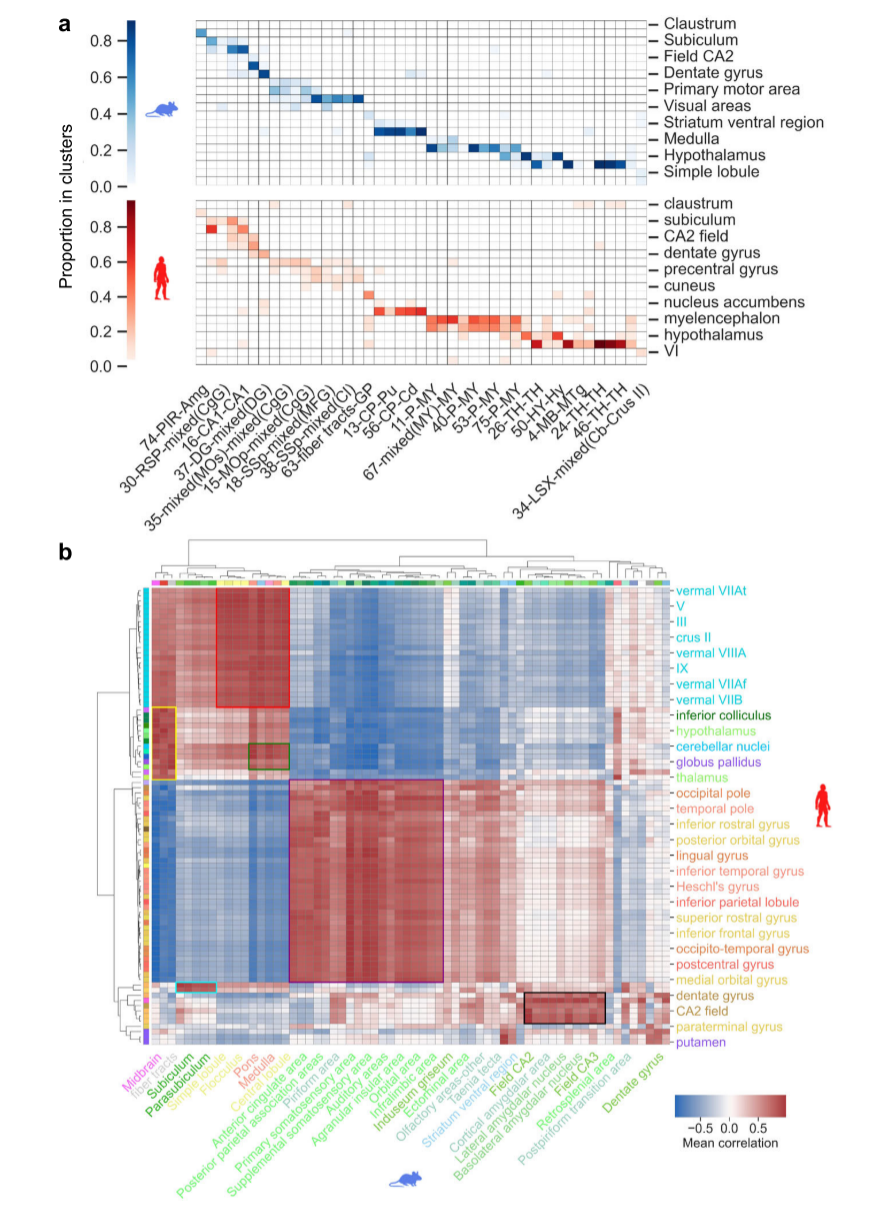
研究人员接下来进行了相关空间转录组分析。首先,他们核查了两个物种聚类群(cluster)和解剖意义上脑区(homologous regions)的对应关系,发现人和鼠的富集情况基本一致,揭示了物种之间基因表达模式的保守性(图3)。
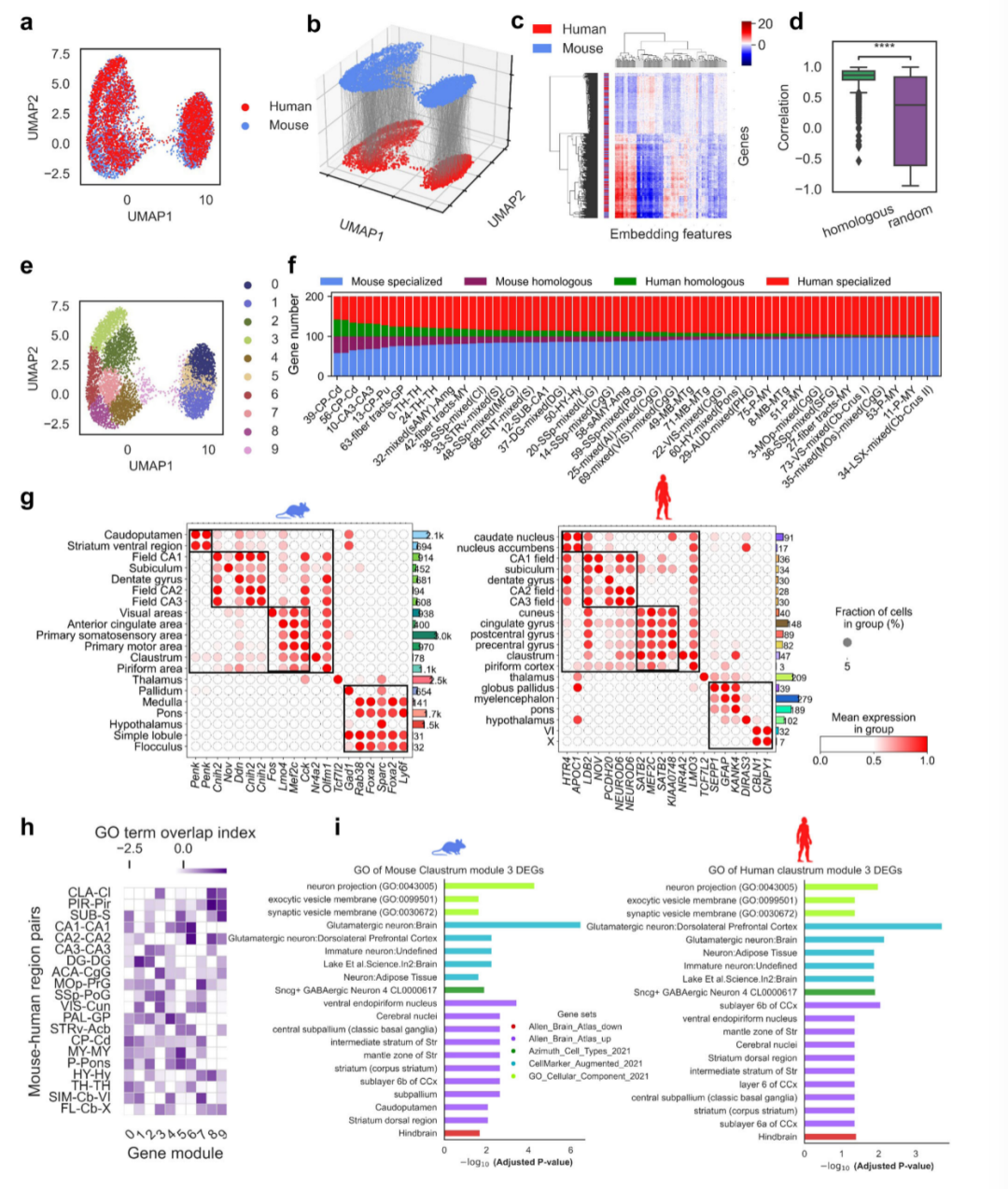
图2和图3均是从测序点(Spots)出发所做的分析,研究人员还从基因(Gene)出发,通过投影到二维UMAP上证明基因的同源关系得到了很好的维护(图4a-d),通过对基因的隐空间嵌入进行聚类,能够发现一些相似的基因表达模块(图4c、e、f)。GO富集分析也显示了人和鼠之间基因富集通路的一致性(图4h-i)。
图4 从基因(Gene)出发揭示跨物种基因表达相似性
最后,研究人员以海马体这一具体组织为例,进一步证实了BrainAlign能够清晰地对齐各个亚群,并证实对应基因也有类似的表达情况(图5)。
总之,在这项工作中,研究者们设计了一个具有自监督对比学习的异构图神经网络来跨物种整合全脑图谱,并通过详细多角度的生物信息学分析证实了跨物种基因表达的相似性。未来融合其他模态数据集(例如,蛋白质相互作用网络)可能会促进更大的图的整合和对齐。
1. Kaas, J. H. The evolution of neocortex in primates. Prog. Brain Res. 195,91–102 (2012).
2. Liu, X., Shen, Q. & Zhang, S. Cross-species cell-type assignment from single-cell RNA-seq data by a heterogeneous graph neural network. Genome Res. 33,96–111 (2023).
董弘禹 | 编译
大模型与生物医学:
AI + Science第二季读书会
生物医学是一个复杂且富有挑战性的领域,涉及到大量的数据处理、模式识别、理论模型建构和实验验证等问题。AI基础模型的引入,使得我们能够从前所未有的角度去观察和理解这个领域的问题,加速科学研究的步伐,提高医疗服务的效率和效果。这种交叉领域的合作,标志着我们正在向科技与生物医学深度融合的新时代迈进,对于推动科学研究、优化医疗服务、促进人类健康有着深远的影响。
集智俱乐部联合西湖大学助理教授吴泰霖、斯坦福大学计算机科学系博士后研究员王瀚宸、博士研究生黄柯鑫、黄倩,华盛顿大学博士研究生屠鑫明,共同发起以“大模型与生物医学”为主题的读书会,共学共研相关文献,探讨基础模型在生物医学等科学领域的应用、影响和展望。读书会已完结,现在报名可加入社群并解锁回放视频权限。
详情请见:
大模型与生物医学:AI + Science第二季读书会启动
拓扑编织着复杂世界,机器学习孕育着技术奇点。一个维度,其中拓扑理论与深度学习模型交织共鸣;一个领域,它跨越了数学的严谨与本质以及人工智能的无限可能,开辟着通往科学新纪元的航道。让我们携手在几何深度学习的起点出发,一路探索如何走向AI for Science的无限未来。
集智俱乐部联合中国人民大学黄文炳副教授、上海交通大学王宇光副教授和南洋理工大学夏克林副教授发起「几何深度学习」读书会。从2024年7月11日开始,每周四19:00-21:00进行,持续时间预计 8-10 周,社区成员将一起系统性地学习几何深度学习相关知识、模型、算法,深入梳理相关文献、激发跨学科的学术火花、共同打造国内首个几何深度学习社区!欢迎加入社区与发起人老师一起探索!
![]()
6. 加入集智,一起复杂!