ChatGPT 理解语言的方式和人类一样吗?卷积神经网络和人脑视觉系统的表征有哪些类似之处?不同信息处理系统之间的差异要如何定量刻画?本文介绍了表征对齐(Representational Alignment)这个跨越认知科学、神经科学、机器学习的通用框架,
并尝试从表征对齐这个角度探讨人工智能和生物智能之间的差异。文章主要介绍了表征对齐是什么,表征对齐的具体步骤,表征对齐所面临的主要技术问题,以及表征对齐的重要性。
研究领域:神经编码,表征对齐,认知计算神经科学,视觉科学,深度学习
荣英淇 | 译述
Raj Magesh | 讨论
陈子睿, 沈泰来, 王志鹏 | 审校
梁金 | 编辑
-
-
-
表征对齐是什么?一个跨认知科学、神经科学、机器学习的通用框架
-
-
-
-
-
-
-
-
-
在人工智能的另一面,人类对生物智能(Natural Intelligence)的探索也从未停歇。我们该如何理解“人会思考”这件事?大脑如何学习新知识?我们如何让自己在学习里有更好的表现?为什么我们能够轻易地做到世界上最先进机器人都不能完成的精细动作?
这篇文章将从表征对齐(Representational Alignment)这个方向探讨人工智能和生物智能的一些差异。我们该如何理解这两种不同的信息处理系统(Information-Processing System)如何接受和处理外部刺激,然后去执行各种任务呢?人工神经网络和生物大脑之间是以同种方式处理或加工信息吗?比如,同样是识别人脸,人和人脸识别软件处理人脸图像的机制一样吗?同样是阅读文章,人和ChatGPT对语言的处理是一样的吗?更广义而言,这里两种信息处理系统可以是两个不同的人,两个不同的人工神经网络,或者是其他执行同样任务的计算模型。
这篇文章将整理部分文章对这些问题进行简述,案例则大多来源于视觉相关研究,所以只能作为一个简单的引子,如果想要更详细探索的老师同学,欢迎直接参阅参考文献。
文章题目:Can an emerging field called ‘neural systems understanding’ explain the brain?
文章链接:https://www.thetransmitter.org/neural-networks/can-an-emerging-field-called-neural-systems-understanding-explain-the-brain/
信息处理系统在处理外部刺激的时候,会根据任务需求的不同采取相应的策略来抽象数据形成内在表征,系统接着根据其内在表征做出相应的行为来完成任务。例如,对于人脑的视觉系统,在完成图像分类、物体识别等各种视觉任务时,从初级视觉区到高级视觉区,对视觉刺激的神经编码也会出现逐渐抽象化的表现,初级视觉区是对图像的初级信息(如边界探测、空间信息)的表征,而高级视觉区会更多地反映图像的语义信息,和物体识别等功能更相关 [1-3]。
注1:有些研究者宣称表征一定要有明确的物理或功能含义,但有些研究者认为表征不具有具体含义,但我们可以用一个高维矢量,矩阵或张量来表示即可。在后文所阐述的对齐背景下,我们往往采纳后者的定义。
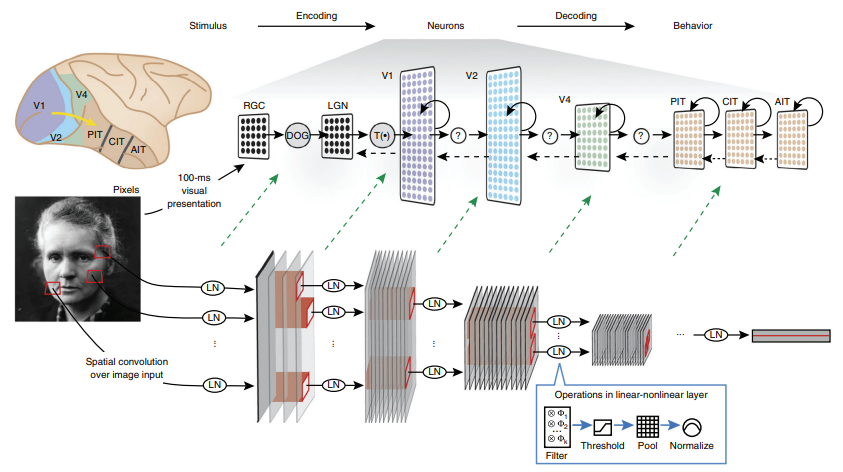
了解计算机视觉的朋友会发现,人工神经网络尤其是卷积神经网络(Convolutional Neural Network, CNN)在视觉任务中有类似的处理机制(这也是受了人脑初级视觉系统的启发)[4]。卷积神经网络的前层大部分是通过卷积操作来完成类似于边界探测的功能,进而对图像的初级信息进行表征,而后层则会出现一些更抽象和难以理解的表征(见图1)。这时候,自然而然会出现一个问题,当我们直观上了解卷积神经网络和人脑视觉系统的表征有类似之处,该如何用定量方法去衡量这种相似程度呢?这就是属于表征对齐大框架下的相关工作。
接下来让我们详细介绍一下表征对齐大框架下的基本步骤,然后提供一个相对更广义的人工神经网络(含不同架构,不同训练任务,以及在不同的训练集训练)与人脑视觉系统对比的案例。
图1: 人工神经网络对图像的表征的一个案例。我们可以发现其第一层的表征具有可解释性,可以明显看到猫的轮廓之类的图像,而在第十层则会变得更加难以解释。其中前层的功能是和如V1之类的初级视觉区类似。|图源:What Is ChatGPT Doing … and Why Does It Work?—Stephen Wolfram Writings
3. 表征对齐是什么?
一个跨认知科学、神经科学、机器学习的通用框架
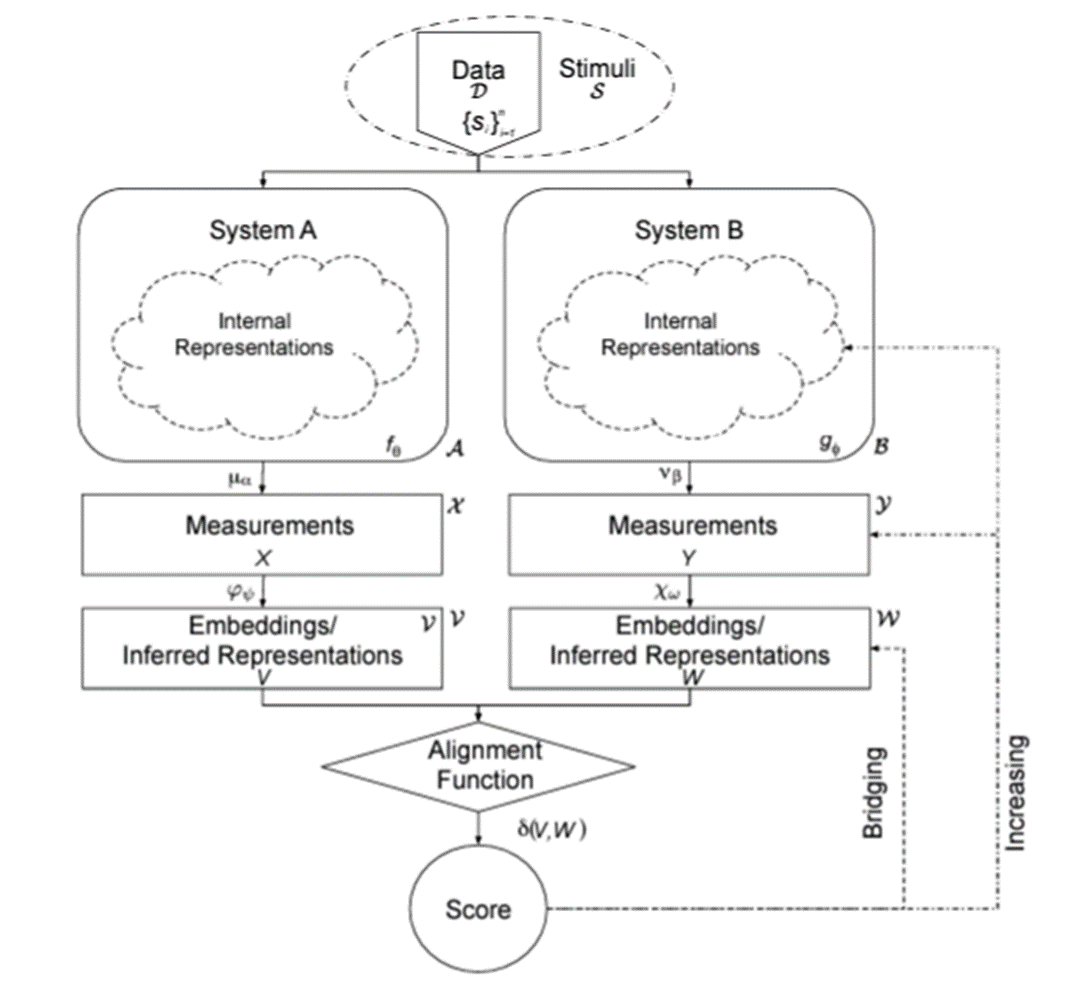
a. 表征对齐的具体步骤及重要元素[5](见图2)
图2:表征对齐的基本步骤和挑战。无论讨论认知科学、神经科学还是机器学习下的表征对齐,大体脱不了这个抽象框架。其中每个实箭头所对应的函数都代表一种统计上的可能操作和选择,虚箭头指向的是表征对齐所面临的技术问题。|图源[5]。
(1)对齐所使用的数据(Data)。对于任何外部刺激(Stimuli),我们可以通过采样其中有限个点来形成子集作为数据去做对齐。比如,对于无限的像素组合,我们可以在其中选取一些与研究问题相关的图像集合;
(2)对齐所选取的系统(System)。当系统接收到对齐使用的数据,其会相应地对数据产生内在表征(Internal Representation)。比如,将图像输入某类人工神经网络中或展示给实验人员后,神经网络整体上对图像的响应,人脑视觉皮层对图像的内在表征;
(3)对系统内在表征的测度(Measurements)。我们需要打开系统对其内在表征进行测度。比如,人工神经网络单独一层的激活,用核磁共振(functional Magnetic Resonance Imaging, fMRI)去衡量视觉皮层的响应。或者我们直接希望人工神经网络或实验人员基于图像做出行为决策 [6-7](这篇文章没有包含不同系统在行为表征的对齐,感兴趣的朋友可以翻看此处的引用,与视觉或语言表征的可解释性更高度关联,或者回顾[5]会有更多不同子领域的案例);
(4)从测度变成嵌入(Embeddings)。进行测度后,我们还需要将其投射于高维连续矢量空间以形成嵌入,这些嵌入可能会有更高的信噪比,或者因为一些工程问题我们需要进行这份操作(比如计算资源有限的时候需要进行降维)来进行对齐的工作。比如,对人工神经网络的激活或脑数据进行降维,将离散的分类数据通过算法映射到高维矢量;
(5) 用来衡量嵌入之间对齐度所选取的对齐函数(Alignment Function)。将得到的嵌入通过对齐函数进行对比,得到相关数值。比如,在机器学习领域常用的 CKA(Centered Kernel Analysis)去衡量模型表征的相似度[8]。
b. 从案例了解具体步骤(见图3)
在[9]中研究者发现,猕猴的初级视觉区的神经活动能够被卷积神经网络的前层更好地预测,而高级视觉区的活动则能够被后层预测;且相比于之前很多设计出来专门预测大脑表征的计算模型,自下而上训练出来只用来分类图像的模型对大脑表征的预测能力更好,可见图2。这个发现启发了后续很多相关工作,众多研究者试图寻找哪种神经网络或其他广义的计算模型能够产生更类脑的视觉表征(更高的brain score)[10]。
图3:同一张图像(玛丽居里)被输入进猕猴的视觉皮层以及卷积神经网络,我们可以发现层与层之间的表征对齐。神经网络中层的响应和中级视觉区V4的响应对齐度较高,神经网络后层的响应与高级视觉区IT的响应对齐度较高。图源[18]
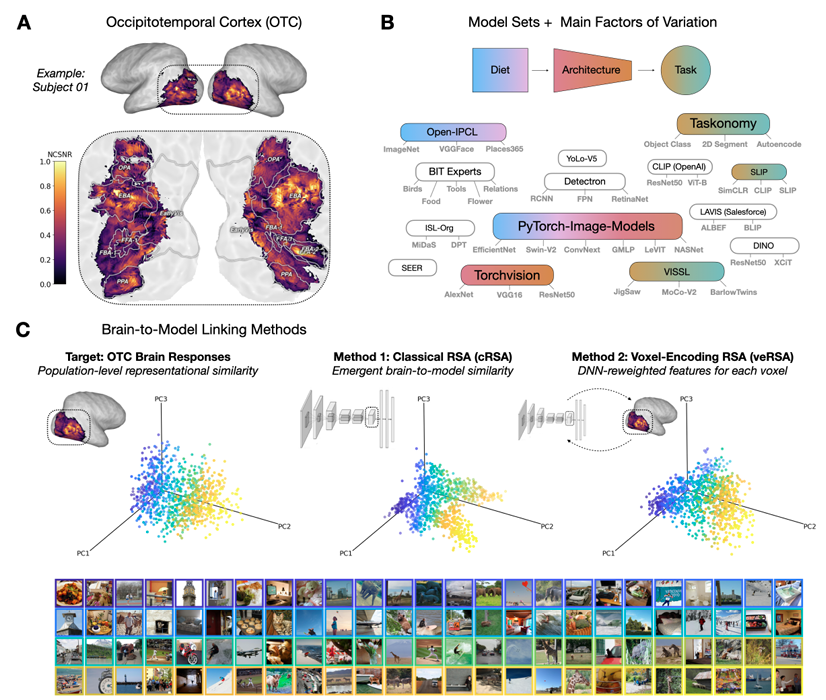
在[11]中,研究者比较了超过200个不同的视觉神经网络。这里,让我们回顾具体步骤来理解这项研究:
(1)广义的外部刺激是视觉刺激,数据涵盖1000张从公开数据集 NSD(Natural Scene Dataset)中被所有实验人员看到的图像[12];
(2) 对于人工神经网络这个系统,我们使用不同模型所对应的预处理图像,然后将其输入形成表征;对于实验人员的人脑,NSD 的研究人员在核磁共振的环境下向实验人员去展示和学习图像,只让他们做一个简单的实验任务:你在之前是否看到过这一图像?每个实验人员都会三次看到同样的图像;
(3)研究者将人工神经网络所有层的激活都当成模型表征的测度,将NSD中fMRI(功能核磁共振)所测量的代表高级视觉区的 OTC(Occipitotemporal Cortex)的反应对图像去做人脑表征的测度;
(4)考虑到计算资源和不同层神经网络的维度可能有所差异,研究者使用稀疏随机投影(Sparse Random Projection)让每层的激活都投射到同一维度上形成模型对图像表征的嵌入[13]。对人脑 OTC 则是将三次不同试验的 fMRI 数据进行平均来当嵌入,这时候的数据不会取决于具体的试验情况而更能反映 OTC 对图像本身的表征;
(5)研究者选取了两种不同的对齐函数去确保其计算的表征对齐度不受制于某种对齐函数的特性,具体的统计处理细节欢迎大家参照原文[14-17]。最后展示的对齐度是基于人工神经网络单独层对 OTC 的最优对齐的那个数值。
最后,研究者发现,不管框架或者训练任务的不同,其与人脑的视觉表征都会有相对一致的对齐度,神经网络训练所使用的图像集对表征对齐度产生影响,详细结论请参照原文[11]。
图4:A. OTC人脑所在区域;B. 选取的模型类型,在模型训练集(Diet)、 架构(Architecture)以及训练任务(Task)上有变化;C. 研究所使用的对齐函数以及选取的图像刺激案例。图源为[11]
c. 表征对齐所面临的三个技术问题(见图2)
在了解表征对齐的基本步骤后,我们将表征对齐所要面对的挑战分为三类:
(1)测量(Measuring)。主要问题在于如何选取对齐函数来计算表征对齐的数值。有关对齐函数之间的对比可见[5, 19]。比如说,我们可以通过设计争议性刺激(Controlversial Stimuli)去生成能最大化区分不同模型的图像,而不是随机选取自然图像来作为对齐所使用的数据,因此形成更强的对齐数指标来区分模型[20]。在脑与模型的比较中,对齐函数的最新发展和相关讨论可以参见:BOTBM3, Battle of the Brain-Model-Mapping Metrics(https://botbm3.github.io/talkseries#outline)。
(2)桥接(Bridging)。主要问题在于如何将不同系统的表征空间投射到同一空间中。一个具体案例是在[21]中,研究者试图通过超对齐(Hyperalignment)寻找不同人之间视觉表征的共同编码(common code)。
(3)提升(Increasing)。主要问题在于如何通过约束或改变系统的表征,让其与另一个系统的对齐程度提升。相关案例是,机器学习研究者训练学生网络(Student Network),使其表征更加接近规模更大、训练成本更高、性能也更好的老师网络(Teacher Network)[22-24]。
a. 一些相关问题
在基本了解表征对齐做什么以后,读者可能会进一步追问,这又意味着什么呢?表征对齐值的高低意味着什么呢?从一些具体问题来讲:
(1)表征对齐值背后的信息含义是什么?其特征是否具有重要含义?
反例:大语言模型(Large Language Model, LLM)能够很好地预测人类对语言处理的神经编码表征,但同样的话我们可以只用类似于句子长度或句子位置这些琐碎的特征,就可以解释大模型语言表征对人脑语言表征的大部分预测效果[25]。这种情况下,表征对齐值哪怕很高却也没能提供很多有趣的信息。
中例:当我们回顾表征对齐的五个步骤时,会发现有许多需要选择操作的地方,数据该如何选取,通过什么样的实验流程将数据输入系统,以什么方式去测量系统的内在表征,选择怎样的嵌入方式,最后又是怎样的对齐函数,都存在很多操作空间。研究者应该意识到对结果的解读都应该建立在这些操作上,因为操作方式可能存在极大的差异,对不同研究结果进行对比时需要谨慎。
正例:在图像处理中,神经网络的前层可以与低级视觉区对齐,而后层可以与高级视觉区对齐。对于这个结果,也有许多相关的迁移应用,比如在考虑高阶视觉功能如识别社会交互的时候,会把神经网络的前层作为一个变量去除视觉低阶表征的影响[26],或者将神经网络的后层作为高阶表征,用来表示自上而下(top-down)的视觉信号来替代预测编码(Predictive Processing)的影响[27]。
(2)我们能够通过计算出神经网络之间的表征对齐值来识别不同的神经系统吗?如果我们发现A网络的表征更接近于B网络而非C网络,我们可以断定B网络的性质(如结构)会和A网络更趋同吗?
结果:不能。如果区分都做不到的话,为什么研究者期待通过表征对齐值能够对神经系统的内在性质有很好的解读呢?[28]
(3)如果相较于其他神经网络,一个神经网络对图像的表征更接近人脑对图像的表征,我们应该期望这个神经网络具有什么样的性质呢?
反例:无论架构、训练任务和模型表现有多么不同,当我们比较超过200个视觉任务下的神经网络,其与大脑的表征对齐程度是一致的。因此,我们很难断定与脑的表征对齐程度对模型本身的理解有什么帮助[11]。
正例:从神经正则化(Neural Regularization)的角度出发,会有很多相关研究,比如具有更类脑表征的神经网络会展现出更强的鲁棒性(Robustness)[29-30];
希望上述的一些案例能够让读者认识到,我们需要对表征对齐的结果有审慎的解读,不过分迷信但也关注潜在应用。接下来会具体从维度视角讨论表征本身为什么重要,以及简述一些表征对齐的案例。
b. 从维度视角来看表征为什么重要
关于表征为什么重要的讨论相关文献汗牛充栋,比如在[31]中,我们可以问表征是否依赖具体的坐标系统,此处只提供一个很窄的视角——维度(Dimensionality)[32],案例也多聚焦于视觉的脑神经编码。
从维度视角出发,表征的意义大致可以分为两类,(1)表征维度的统计结构,例如高阶认知是否需要通过高维表征如混合编码(Mixture Selectivity)来实现 [33-35];(2)表征维度的可解释性,例如能否通过独立成分分析(Indepdent Component Analysis, ICA)去理解哪些特征构成了表征。
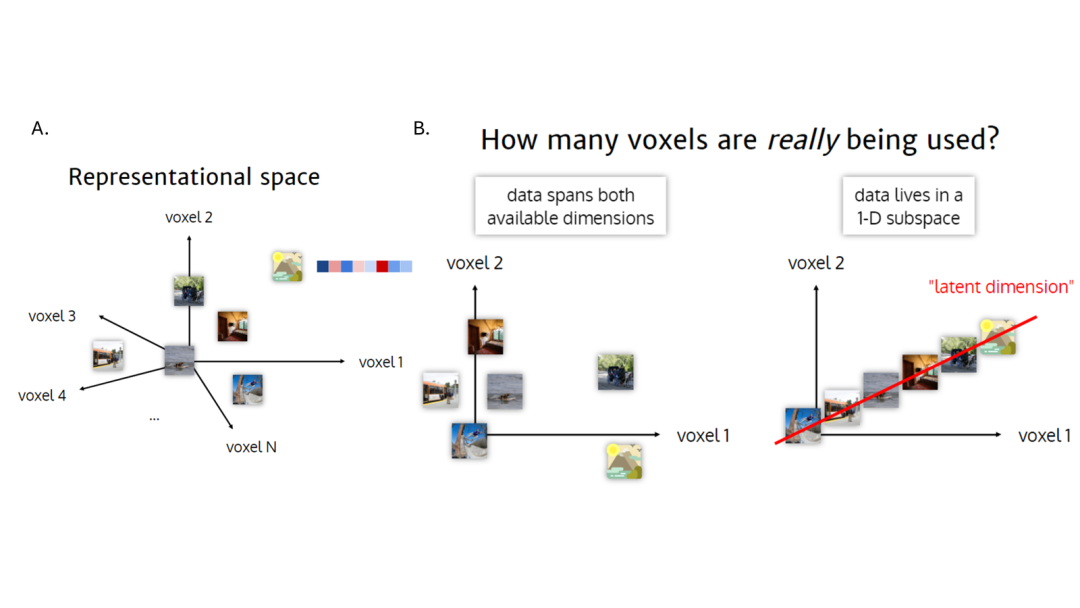
我们可以从“视觉皮层对图像刺激的表征是高维的还是低维的”这个具体问题出发,来理解这两层意义。图5中简单地说明了维度对视觉表征意味着什么。
图5:A. 示意图,脑的神经编码如何表征不同的自然图像;B. 示意简化图,当表征空间是二维的时候,左图意味着我们需要两个维度去表征自然图像,右图意味着我们只需要一个维度去表征自然图像。图源:https://slides.com/raj-magesh/scale-free-visual-cortex/
在这一问题下,其中一个比较基本的统计方法是主成分分析(Principal Component Analysis, PCA)。
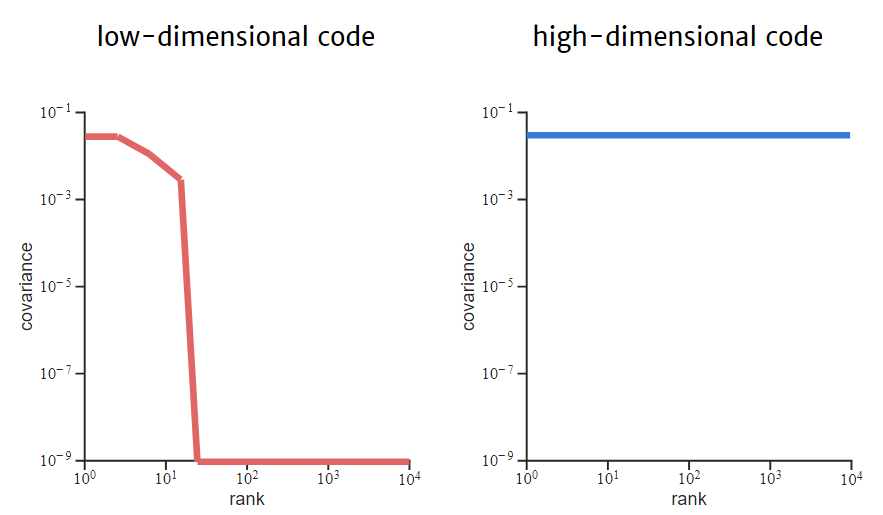
如果视觉皮层的神经编码(Neural Code)每个维度都编码单个特征,其效率(Efficiency)是最高的。当我们对此类表征进行主成分分析的时候,会发现其特征谱(Eigenspectrum)往往是条平线,这表示我们需要所有的维度来去解释表征空间的方差(Variance),便意味着高维度(见图6右)。而对于低维的神经编码,当我们对其表征进行主成分分析的时候,特征谱的方差会很快下降然后趋近于或者变成0(见图6左)。
当我们解读视觉皮层对自然图像的表征时,会发现其往往是一条方差逐步下降趋近于0的曲线,许多研究常用的方法是选取前面少量最重要的维度,一般来说可能选取数量不多于10个,也有部分研究会选几十个,去宣称视觉皮层对图像的信息表征就在这些维度里面。而做可解释的研究人员会试图解释这些主成分维度,认为前几个主成分维度的图像可能是脸部特征、食物或者活物,这些可能是对脑神经编码最重要的信息。
图6:左图是低维的神经编码的特征谱,右图为高维的神经编码的特征谱。y轴代表的是维度所能解释的数据方差,x轴代表的是按方差排序从高到低的隐性维度。图源:https://slides.com/raj-magesh/scale-free-visual-cortex/
那为什么依然有不少研究者认为视觉神经编码表征(当其特征谱是逐渐下降的曲线时)是高维的呢?虽然视觉神经编码表征的特征谱不是条平坦的线,但如果我们将特征谱放入对数刻度(log-log scale)中,会发现幂律(Power Law)出现了。这意味着虽然后续的维度对表征方差的解释极低,但其似乎不能被当成噪声来处理。在[36]中,研究者宣称这种符合幂律的高维表征,是因为表征也需要平滑性(Smoothness)这个约束(平滑性能够保证,当我们对系统输入进行小扰动时,系统输出不会出现剧烈变化。比如,我们会希望在处理两张极为相似的猫的图片时,大脑形成的内在表征不会有很大差异)而不能仅仅只追求高效率。甚至他们在小鼠钙信号的分析中,宣称幂律这条线的斜率都具有一定确定性,因为其刚好满足效率和平滑性的最佳平衡。有一些人工神经网络的研究也受到了这一发现的影响,在训练中增加了表征呈现幂律特征的约束,但其如何影响神经网络的性能还存在争议[37]。
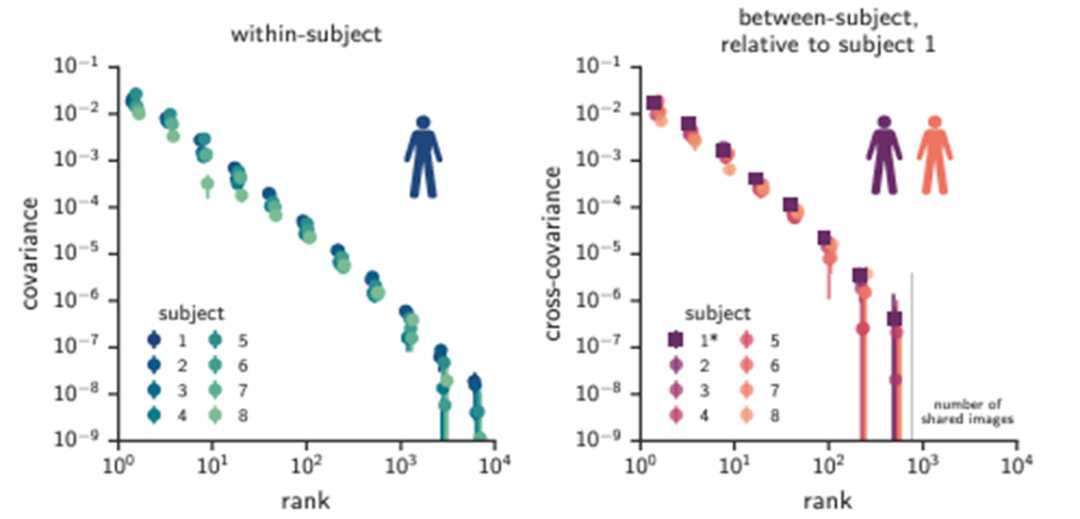
在[38]中,研究者进一步发展了上述研究所使用的统计手段(不是传统的PCA, 关于计算方法的教程可见[39]),其在一定程度上在做PCA的同时能确保噪声维度的可解释方差(Explained Variance)为0。研究者通过计算同一实验人员跨实验或者不同实验人员共有神经编码信息的特征谱,发现视觉皮层的高维表征是一个不局限于具体试验或者具体实验人员的通用属性(见图7),且在人的不同脑区保持一致。在后续研究中,当研究者用同样的方法分析小鼠的数据,同样的幂律会在两种不同的神经信号测量方法(核磁共振和钙成像)都存在。当研究者进一步采取粗粒化手段去缩放神经信号的空间尺度,发现这种高维表征依旧存在[40]。这时候,如果我们再试图讨论维度的可解释性对于更高的维度来说,似乎便很难找到明确的意义了。当然,哪怕同样是支持视觉表征是高维且符合幂律的学者,其中也有不少争议[41]。
图7:左图是不同实验人员跨试验视觉响应区的神经编码的特征谱,右图是跨实验人员之间视觉响应区的特征谱,会发现都符合幂律的特征,图源为[38]。
综上所述,从维度角度出发,表征的重要性可以体现成两方面:(1)从统计结构出发,表征是高维的还是低维的,其特殊性质可以反映信息处理系统的机制,例如:脑对视觉刺激的高维表征是需要在效率和平滑性得到平衡,另外更高维的人工神经网络表征可以得益于其更高的效率和表达性(Expressivity),因此对脑表征的预测以及新任务学习有所助益[42];(2)从可解释性(Interpretability)出发,我们可以理解不同的特征对信息处理系统的重要性,例如,人在识别物体的时候形成的表征能被物体是否为活物(animate or not)及其尺寸大小来解释[43]。
此处,我们可以介绍两个有关表征的研究,似乎可以说明表征本身的性质在不同的系统下可能有很强的共通性,因此可能意味更有趣的性质。
c. 柏拉图表征(Platonic Representation)[44]
(洞穴假说版本)随着规模的增长,无论架构、算法还是训练任务,神经网络(大语言模型、视觉神经网络等)的表征会趋于相似,且意味着更高的模型表现,似乎印证了洞穴假说中的理想表征;
(安娜卡列尼娜版本)好的神经网络表征都是相似的,而不好的神经网络表征各有各的不同。
注2:在笔者的个人成长历程中,柏拉图的洞穴假说启蒙了我对哲学的好奇心,《安娜·卡列尼娜》则是文学里的最爱。而这两种元素竟然都会在同一篇名为柏拉图表征猜想的论文被提及,真是莫大的惊喜。
在研究者发现这个现象的同时(当然这个现象本身是否有坚实的统计基础,欢迎感兴趣的朋友去进一步探索),研究者也简单地阐述了这种现象背后的可能成因。
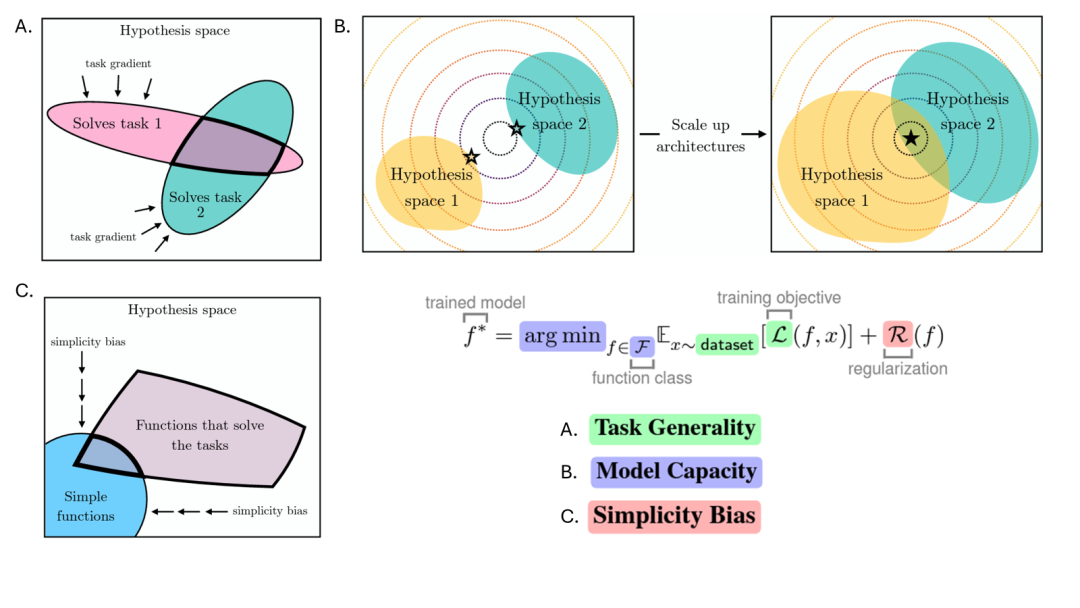
(1)随着神经网络所需要执行的任务越来越多,也越来越困难,其参数所在的可选择空间也越来越小(见图8A);
(2)如果存在一个最优表征,规模越大参数越多的模型更容易找到这个表征,而不是陷入最优的局部解(见图8B);
(3)神经网络训练时的正则化会鼓励不同的神经网络在所有可能拟合数据的方式里找到相对简单的答案(见图8C)。
图8:随着规模增大能力增强,神经网络表征会逐渐对齐的原因猜想。A. 随着人工神经网络需要解决更多的任务,能适应这任务的可能表征范围在缩小。粉色椭圆代表能解决任务1的表征空间,绿色椭圆代表能解决任务2的表征空间,如果一个神经网络要同时解决两个任务,其表征空间只能在椭圆交界的地方;B. 如果最优表征空间存在的话,规模越大的神经网络能搜寻到的表征空间(黄色或绿色椭圆)更有可能找到最优表征的解(黑色五角星)而不是局部解(白色五角星);C. 正则化会让模型寻找拟合数据解的较简解。紫色四边形代表能解决人物的函数,蓝色椭圆代表函数要相对简单,正则化会希望找到的表征空间是其交界。图源为[44]。
如果这个简单的猜想是正确的,那我们是否期待理想表征会出现呢?其和生物大脑的表征又有什么关系?我们该期待它们之间有关系吗?
d. 通用维度(Universal Dimension)[45]
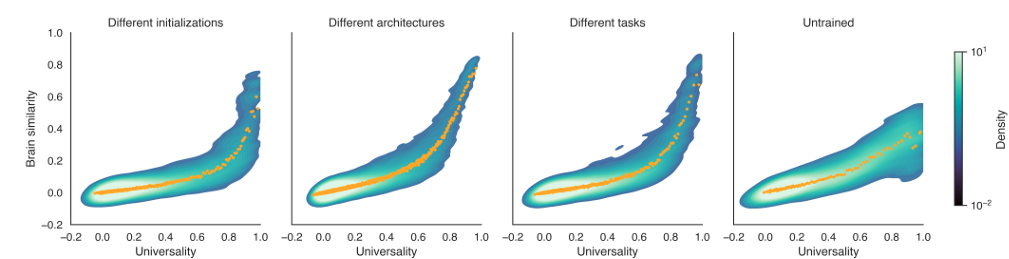
研究者主要关注的是人工神经网络和人脑视觉皮层对自然图像刺激的表征对齐。给定一组人工神经网络的表征,我们可以将所有信息都拆分成不同的维度,接下来我们可以分析单个维度被这组内多少的神经网络共享,如果这个数量较高,我们可以认为这个维度是在很多的神经网络都存在的,或者可以被理解为通用性(Universality)较高。研究发现,人工神经网络表征维度的通用性和其与人脑视觉皮层的对齐度是正相关的(见图9)。
图9:人工神经网络表征中维度的通用性和其与对人脑视觉表征的对齐成正相关,从左到右是不同初始化、不同架构、不同任务以及未经训练的神经网络进行分析。图源为[45]
这篇文章对表征对齐这个领域有个简要梳理,如果希望有更深入理解的欢迎直接阅读[5],读者也可以通过以下的问题清单去梳理自己所得:
1. 表征对齐的基本步骤是什么?能否举出一个具体案例;
2. 如果我们说两个系统的表征接近,可以说明这两个系统的其他内在性质(例如,对人工神经网络而言,其架构,任务目标和学习算法)会有更高的相似性吗?
3. 当我们讨论表征的维度时,可能在讨论什么?
4. 脑神经系统对自然图像的视觉表征是高维的还是低维的?
5. 什么是柏拉图表征?其可能和脑表征的关联是什么?如果我们要质疑其结果的有效性,有哪些可能的方向?
注3:这篇文章远远没有涵盖相关领域的发展,感兴趣的朋友欢迎自行阅览参考文献和找到更多有趣相关的其他论文,比如对于人工神经网络如何能帮助我们解决神经科学问题,可参见相关文章[18,46-52]。
[1] Hubel, D. H., & Wiesel, T. N. (1962). Receptive fields, binocular interaction and functional architecture in the cat’s visual cortex. The Journal of physiology, 160(1), 106.
[2] Olshausen, B. A., & Field, D. J. (1996). Emergence of simple-cell receptive field properties by learning a sparse code for natural images. Nature, 381(6583), 607-609.
[3] DiCarlo, J. J., Zoccolan, D., & Rust, N. C. (2012). How does the brain solve visual object recognition?. Neuron, 73(3), 415-434.
[4]Lindsay, G. W. (2021). Convolutional neural networks as a model of the visual system: Past, present, and future. Journal of cognitive neuroscience, 33(10), 2017-2031.
[5]Sucholutsky, I., Muttenthaler, L., Weller, A., Peng, A., Bobu, A., Kim, B., … & Griffiths, T. L. (2023). Getting aligned on representational alignment. arXiv preprint arXiv:2310.13018.
[6] Mahner, F. P., Muttenthaler, L., Güçlü, U., & Hebart, M. N. (2024). Dimensions underlying the representational alignment of deep neural networks with humans. arXiv preprint arXiv:2406.19087.
[7] Du, Changde, et al. “Human-like object concept representations emerge naturally in multimodal large language models.” arXiv preprint arXiv:2407.01067 (2024).
[8] Kornblith, S., Norouzi, M., Lee, H., & Hinton, G. (2019, May). Similarity of neural network representations revisited. In International conference on machine learning (pp. 3519-3529). PMLR.
[9] Yamins, D. L., Hong, H., Cadieu, C. F., Solomon, E. A., Seibert, D., & DiCarlo, J. J. (2014). Performance-optimized hierarchical models predict neural responses in higher visual cortex. Proceedings of the national academy of sciences, 111(23), 8619-8624.
[10] Schrimpf, M., Kubilius, J., Hong, H., Majaj, N. J., Rajalingham, R., Issa, E. B., … & DiCarlo, J. J. (2018). Brain-score: Which artificial neural network for object recognition is most brain-like?. BioRxiv, 407007.
[11] Conwell, C., Prince, J. S., Kay, K. N., Alvarez, G. A., & Konkle, T. (2022). What can 1.8 billion regressions tell us about the pressures shaping high-level visual representation in brains and machines?. BioRxiv, 2022-03.
[12] Allen, E. J., St-Yves, G., Wu, Y., Breedlove, J. L., Prince, J. S., Dowdle, L. T., … & Kay, K. (2022). A massive 7T fMRI dataset to bridge cognitive neuroscience and artificial intelligence. Nature neuroscience, 25(1), 116-126.
[13] Pedregosa, F., Varoquaux, G., Gramfort, A., Michel, V., Thirion, B., Grisel, O., … & Duchesnay, É. (2011). Scikit-learn: Machine learning in Python. the Journal of machine Learning research, 12, 2825-2830.
[14] Kriegeskorte, N., Mur, M., & Bandettini, P. A. (2008). Representational similarity analysis-connecting the branches of systems neuroscience. Frontiers in systems neuroscience, 2, 249.
[15]Khaligh-Razavi, S. M., Henriksson, L., Kay, K., & Kriegeskorte, N. (2017). Fixed versus mixed RSA: Explaining visual representations by fixed and mixed feature sets from shallow and deep computational models. Journal of Mathematical Psychology, 76, 184-197.
[16]Kaniuth, P., & Hebart, M. N. (2022). Feature-reweighted representational similarity analysis: A method for improving the fit between computational models, brains, and behavior. NeuroImage, 257, 119294.
[17]Konkle, T., & Alvarez, G. A. (2022). A self-supervised domain-general learning framework for human ventral stream representation. Nature communications, 13(1), 491.
[18]Yamins, D. L., & DiCarlo, J. J. (2016). Using goal-driven deep learning models to understand sensory cortex. Nature neuroscience, 19(3), 356-365.
[19] Soni, A., Srivastava, S., Khosla, M., & Kording, K. P. (2024). Conclusions about Neural Network to Brain Alignment are Profoundly Impacted by the Similarity Measure. bioRxiv, 2024-08.
[20] Golan, T., Raju, P. C., & Kriegeskorte, N. (2020). Controversial stimuli: Pitting neural networks against each other as models of human cognition. Proceedings of the National Academy of Sciences, 117(47), 29330-29337.
[21] Haxby, J. V., Guntupalli, J. S., Connolly, A. C., Halchenko, Y. O., Conroy, B. R., Gobbini, M. I., … & Ramadge, P. J. (2011). A common, high-dimensional model of the representational space in human ventral temporal cortex. Neuron, 72(2), 404-416.
[22] Hinton, G. (2015). Distilling the Knowledge in a Neural Network. arXiv preprint arXiv:1503.02531.
[23] Phuong, M., & Lampert, C. (2019, May). Towards understanding knowledge distillation. In International conference on machine learning (pp. 5142-5151). PMLR.
[24] Tian, Y., Krishnan, D., & Isola, P. (2019). Contrastive representation distillation. arXiv preprint arXiv:1910.10699.
[25] Feghhi, E., Hadidi, N., Song, B., Blank, I. A., & Kao, J. C. (2024). What Are Large Language Models Mapping to in the Brain? A Case Against Over-Reliance on Brain Scores. arXiv preprint arXiv:2406.01538.
[26] McMahon, E., Bonner, M. F., & Isik, L. (2023). Hierarchical organization of social action features along the lateral visual pathway. Current Biology, 33(23), 5035-5047.
[27] Richter, D., Kietzmann, T. C., & de Lange, F. P. (2023). High-level prediction errors in low-level visual cortex. bioRxiv, 2023-08.
[28]Han, Y., Poggio, T. A., & Cheung, B. (2023, July). System identification of neural systems: If we got it right, would we know?. In International Conference on Machine Learning (pp. 12430-12444). PMLR.
[29]Li, Z., Brendel, W., Walker, E., Cobos, E., Muhammad, T., Reimer, J., … & Tolias, A. (2019). Learning from brains how to regularize machines. Advances in neural information processing systems, 32.
[30]Shao, Z., Ma, L., Li, B., & Beck, D. M. (2024). Leveraging the Human Ventral Visual Stream to Improve Neural Network Robustness. arXiv preprint arXiv:2405.02564.
[31]Khosla, M., Williams, A. H., McDermott, J., & Kanwisher, N. (2024). Privileged representational axes in biological and artificial neural networks. bioRxiv, 2024-06.
[32]Roads, B. D., & Love, B. C. (2024). The Dimensions of dimensionality. Trends in Cognitive Sciences.
[33]Rigotti, M., Barak, O., Warden, M. R., Wang, X. J., Daw, N. D., Miller, E. K., & Fusi, S. (2013). The importance of mixed selectivity in complex cognitive tasks. Nature, 497(7451), 585-590.
[34]Fusi, S., Miller, E. K., & Rigotti, M. (2016). Why neurons mix: high dimensionality for higher cognition. Current opinion in neurobiology, 37, 66-74.
[35]Tye, K. M., Miller, E. K., Taschbach, F. H., Benna, M. K., Rigotti, M., & Fusi, S. (2024). Mixed selectivity: Cellular computations for complexity. Neuron.
[36] Stringer, C., Pachitariu, M., Steinmetz, N., Carandini, M., & Harris, K. D. (2019). High-dimensional geometry of population responses in visual cortex. Nature, 571(7765), 361-365.
[37]Ghosh, A., Mondal, A. K., Agrawal, K. K., & Richards, B. (2022). Investigating power laws in deep representation learning. arXiv preprint arXiv:2202.05808.
[38]Gauthaman, R. M., Ménard, B., & Bonner, M. F. (2024). Universal scale-free representations in human visual cortex. arXiv preprint arXiv:2409.06843.
[39]Gauthaman, Raj Magesh, Florentin Guth, Atlas Kazemian, Zirui Chen, and Michael Bonner. 2023. “A High-Dimensional View of Neuroscience.” August 26, 2023. https://BonnerLab.github.io/ccn-tutorial//.
[40] Gauthaman, R. M., Ménard, B., & Bonner, M. F. Universality in mouse and human visual cortex: relating covariance to the spatial structure of latent dimensions.
[41]Pospisil, D. A., & Pillow, J. W. (2024). Revisiting the high-dimensional geometry of population responses in visual cortex. bioRxiv, 2024-02.
[42]Elmoznino, E., & Bonner, M. F. (2024). High-performing neural network models of visual cortex benefit from high latent dimensionality. PLOS Computational Biology, 20(1), e1011792.
[43]Long, B., Yu, C. P., & Konkle, T. (2018). Mid-level visual features underlie the high-level categorical organization of the ventral stream. Proceedings of the National Academy of Sciences, 115(38), E9015-E9024.
[44]Huh, M., Cheung, B., Wang, T., & Isola, P. (2024). The platonic representation hypothesis. arXiv preprint arXiv:2405.07987.
[45]Chen, Z., & Bonner, M. F. (2024). Universal dimensions of visual representation. arXiv preprint arXiv:2408.12804.
[46]Richards, B. A., Lillicrap, T. P., Beaudoin, P., Bengio, Y., Bogacz, R., Christensen, A., … & Kording, K. P. (2019). A deep learning framework for neuroscience. Nature neuroscience, 22(11), 1761-1770.
[47]Doerig, A., Sommers, R. P., Seeliger, K., Richards, B., Ismael, J., Lindsay, G. W., … & Kietzmann, T. C. (2023). The neuroconnectionist research programme. Nature Reviews Neuroscience, 24(7), 431-450.
[48]Saxe, A., Nelli, S., & Summerfield, C. (2021). If deep learning is the answer, what is the question?. Nature Reviews Neuroscience, 22(1), 55-67.
[49]Kanwisher, N., Khosla, M., & Dobs, K. (2023). Using artificial neural networks to ask ‘why’ questions of minds and brains. Trends in Neurosciences, 46(3), 240-254.
[50]Kriegeskorte, N., & Douglas, P. K. (2018). Cognitive computational neuroscience. Nature neuroscience, 21(9), 1148-1160.
[51]Yang, G. R., & Wang, X. J. (2020). Artificial neural networks for neuroscientists: a primer. Neuron, 107(6), 1048-1070.
[52]Bowers, J. S., Malhotra, G., Dujmović, M., Montero, M. L., Tsvetkov, C., Biscione, V., … & Blything, R. (2023). Deep problems with neural network models of human vision. Behavioral and Brain Sciences, 46, e385.
人类大脑是一个由数以百亿计的神经元相互连接所构成的复杂系统,被认为是「已知宇宙中最复杂的物体」。本着促进来自神经科学、系统科学、信息科学、物理学、数学以及计算机科学等不同领域,对脑科学、类脑智能与计算、人工智能感兴趣的学术工作者的交流与合作,集智俱乐部联合国内外多所知名高校的专家学者发起神经、认知、智能系列读书会第三季——「计算神经科学」读书会,涵盖复杂神经动力学、神经元建模与计算、跨尺度神经动力学、计算神经科学与AI的融合四大模块,并希望探讨计算神经科学对类脑智能和人工智能的启发。读书会已完结,现在报名可加入社群并解锁回放视频权限。
详情请见:计算神经科学读书会启动:从复杂神经动力学到类脑人工智能
神经科学和人工智能领域的多位著名学者近日发表 NeuroAI 白皮书认为,神经科学长期以来一直是推动人工智能(AI)发展的重要驱动力,NeuroAI 领域的基础研究将推动下一代人工智能的进程。文章发表后引发热议:神经科学是否推动了人工智能?未来的人工智能是否需要神经科学?
本着促进神经科学、计算机科学、认知科学和脑科学等不同领域的学术工作者的交流与合作,集智俱乐部联合北京师范大学柳昀哲、北京大学鲍平磊和昌平实验室吕柄江三位研究员共同发起了「NeuroAI」读书会,聚焦在视觉、语言和学习领域中神经科学与人工智能的相关研究,期待能够架起神经科学与人工智能领域的合作桥梁,激发跨学科的学术火花。读书会已完结,现在报名可加入社群并解锁回放视频权限。
![]()
7. 加入集智,一起复杂!
点击“阅读原文”,报名读书会