Cell 长文综述:机器学习如何助力网络生物学
© Argonne National Laboratory
机器学习正在成为现代生物学研究不可或缺的一部分。2018年7月,Cell 杂刊载了一篇由哈佛大学和科罗拉多大学研究人员完成的综述文章,从疾病生物学,药物发现,微生物组研究和合成生物学等方面,讨论了机器学习与网络生物学交叉研究的机遇和挑战。
本文转载自公众号“再创丨Regenesis”,对机器学习简介部分有删减,完整内容请移步《网络生物学与机器学习:什么是机器学习》。合成生物学作为一门跨学科的新兴领域,试图从工程角度理解和构建生命的复杂性,集智俱乐部将持续关注和报道合成生物学领域的前沿进展。
全文约1万字,阅读时间约为20分钟。
论文题目:
Next-Generation machine learning for biological networks
论文作者:
Camacho, D. M., Collins, K. M., Powers, R. K., Costello, J. C., & Collins, J. J
论文地址:
https://www.cell.com/cell/fulltext/S0092-8674(18)30592-0
在过去的十年中,通过对分子变量如基因,蛋白质和代谢物丰度,微生物组成和群体遗传变异等进行量化,生物学领域产生了大量高度复杂的数据集,且数据集的数量正在急剧增加。跨研究学科的研究中经常会产生PB量级的数据。例如,Cancer Genome Atlas从30,000多个不同癌症类型的患者身上采集的数据,总计已经超过2.5 PB。类似项目,如Human Microbiome Project,ENCODE Project Consortium, 以及100,000 Genomes Project 等等也正在产生大量数据。
这些数据集提供了深入了解生物系统和复杂疾病所需的原材料,但如何进一步探索这些数据的价值只能通过更高层次的分析过程来实现。上面说到的项目时刻在向我们表明:处理大型异构复杂数据集的工具和技术正在变得无比重要。
这是机器学习和网络生物学交叉的机遇,这是一个处理生物网络和大型多维数据集的研究领域。机器学习的应用在生物学中变得无处不在,不仅近包括基因组注释,而且还包括对蛋白质结合的预测,癌症关键转录驱动因子的鉴定,复杂微生物群落中代谢功能的预测,和转录调控网络的表征等等。简而言之,任何可以用来学习相应的模式并应用于新数据集的任务都属于机器学习的范畴。机器学习方法一个关键的优势是可以用来筛选大量的数据以探索可能会被忽略的模式。在生物和生物医学研究的大数据时代,机器学习在发现复杂生物系统的可预测模式方面起着关键的作用。
随着我们在生物系统的多个层面上收集到越来越庞大且多样化的数据,我们可以利用这些数据集设计相应的机器学习方法,在多个层面(从基因调控到物种间相互作用)构建更复杂、更真实的网络模型。
网络生物学涉及研究生物分子的复杂相互作用,该领域在生物系统建模中扮演着重要角色,辅之以跨多个多元组学程序生成的高度复杂的数据集。网络生物学涉及大规模内生生物网络(系统生物学背景)的重建和分析,以及小规模合成基因网络(合成生物学背景)的设计和构建。
此外,下一代机器学习方法提供的工具可以增加这些网络模型在各种生物医学领域中的应用。 在接下来的内容中,我们将会从应用综述、疾病生物学,药物开发,微生物组研究和合成生物学这五个方面探讨网络生物学与机器学习的交叉研究将如何在这些领域发挥重要的作用。
机器学习在生物学中的应用
在选择机器学习算法应用于生物学问题时,需要考虑几个因素,特别是考虑到生物数据的可变性以及用于收集数据的不同平台和方法时。由于技术和生物本身的差异,在一个数据集上训练的机器学习模型可能无法很好地推广到其他数据集。任何新的数据集应该与用于训练模型的数据的一般属性相匹配。新数据也应该使用与训练数据相同的流程进行处理。如果新数据与训练数据存在显著不同,机器学习模型的预测很可能是虚假的。
与分子生物学技术非常相似,机器学习方法也是要根据特定情形才能决定的。机器学习和分子生物学实验都需要仔细的实验设计才能正确检验假设。尽管机器学习的目标是开发可以广泛应用的预测模型,但模型根据不同预测条件可能会做出不同的假设,并且其性能可能会在不同条件下发生变化。所有方法的选择都需要作出权衡; 这个概念在计算机科学中是被广泛认可的,称为“No free lunch theorem”。
机器学习的性能可能受多种因素的影响,包括特征选择,用户定义的参数以及方法本身。直接证据表明这些因素是生物应用中影响机器学习性能的主要因素。这些证据可以在Reverse Engineering Assessment and Methodology(DREAM)Challenge中找到。DREAM Challenge旨在寻找网络生物学和医学中大数据研究问题的解决方案。以前的挑战项目包括基因组规模调控网络的推断以及使用多元数据集对药物敏感性和协同作用的预测。许多生物网络推断方法可以被定义为无监督学习,其中输入数据用于在给定一组实验数据集的情况下预测生物分子(特征,feature)之间的相互作用(边缘,edges)。第二类生物网络推理算法使用监督学习方法对新样本进行预测。此类方法在表征药物作用机制或疾病状态驱动因素方面取得了重大的成功。
图2. DREAM Challenge.
链接:http://dreamchallenges.org/
每个DREAM Challenge向网络生物学研究界提出一个具体问题和解决它的必要数据。这些问题需要利用计算模型(通常是机器学习方法)来解决,但对应用的模型类型没有限制。每个挑战都包含一个隐藏的评估数据集,用于评估每种方法的性能,从而提供独立的,无偏见的评估来评估不同的方法。随着几十个挑战已经完成,我们可以大致总结出三条“经验法则”,为机器学习方法在网络生物学中应用提供借鉴:
(1)“简单”通常更好(Simple is often better):不管挑战如何,几乎可以肯定的是,一个简单的机器学习方法将成为表现最佳的模型。这些模型通常包括基于线性回归的模型(例如弹性网络),这些模型在一系列机器学习任务中表现良好,因此具有出色的起点。
(2)先验知识提高性能(Prior knowledge improves performance):领域特定知识的应用几乎总是有助于任何预测模型。例如,使用磷酸蛋白质组学数据来对乳腺癌中的信号传导网络反向工程。信号网络中元件和相互作用的先验知识增强了机器学习预测信号相互作用的能力。
(3)集成模型产生的结果更加稳健(Ensemble models produce robust results):在开始我们讨论过,集成模型整合来自多个独立预测方法的预测。集成模型在各种挑战中一直是最佳的模型选择,并且往往鲁棒性更高。
在不同的DREAM Challenge中,可以看出,没有任何一种机器学习方法或一类方法总是表现最佳。因此,没有“magic bullet”方法可以最优地解决网络生物学中的所有机器学习任务。

疾病生物学
网络生物学可以帮助我们更好地理解疾病的复杂性。 传统方法依赖于疾病特定方面的识别和特征描述,例如疾病相关基因的发现,而网络生物学采取的是更加全面的方法,因此,网络生物学将为我们揭示更加全面的疾病表型驱动因素的信息。 网络生物学不是简单地识别潜在的生物标志物,而是让我们能够对疾病状态中至关重要的生物分子相互作用的网络和子网络进行描述。
在定义疾病的网络特定特征时,可以合理地使用机器学习算法来帮助理解和定义潜在的疾病机制。比如,可以使用来自诸如BioGRID来源的现有生物网络知识,探索与健康状态相比,不同生物分子之间的关系如何在疾病状态中发生变化(BioGRID是一个关于基因相互作用,蛋白质 – 蛋白质相互作用,化学相互作用,翻译后修饰的数据库)。从健康队列的数据开始,我们可以训练深度学习算法(例如,深度神经网络)来学习和定义健康状态的基本特征。训练后,我们可以将来自患者群体的数据提供给算法,并用于预测健康状态和疾病状态之间的差异等应用。
我们需要更好地了解疾病背后生物网络的复杂结构,以及这些网络的失调如何可能导致某种疾病状态。在这方面,胶囊网络(Capsule networks),可能具有很高的应用价值。胶囊网络涉及一种新型的神经网络架构,其中CNN被封装在相互连接的模块中。如前所述,CNN是处理多维数据的一种特殊的深层神经网络,例如网络生物学中发现的组学数据集。另一方面,胶囊网络是将深度神经网络表示为一组模块(胶囊)的代表,其允许以保持数据本身分层结构的方式来学习数据结构。这在图像数据分析中特别有用,因为它允许算法学习独立于图像视角的图像特征。
考虑到生物网络本质上是高度模块化的,胶囊网络已经可以成熟的应用于网络生物学和疾病生物学:胶囊网络为生物分子指定层,同时允许每一层与其他层相互作用。每个生物层可以被视为胶囊。横跨不同生物层产生的数据(例如转录组学,蛋白质组学,代谢组学)可以训练与每个胶囊相关的CNN从而独立地了解这些层中的特定性质。在胶囊之间应用动态路由(Dynamic routing)的前提将允许不同胶囊将任何其他胶囊的输出作为输入,从而使模型能够了解每个层之间的相互作用和依赖性。这种方法将允许人们研究高度模块化的系统,如由基因,蛋白质,代谢物等组成的生物网络,并分析这些网络及其子网络的功能组织和相互作用在疾病状态如何被破坏。
我们并没有注意到胶囊网络的任何生物学应用,但它们的独特特征可以使我们能够解开和解决人类疾病的复杂性。 正如我们下面所描述的那样,胶囊网络和其他深度学习方法的成功实施将取决于适当大、高质量、注释良好的数据集的可用性。
药物开发
在药物开发中,我们迫切需要表征化合物的作用方式、识别药物的脱靶效应并开发有效的药物组合来治疗复杂疾病。 网络生物学以及机器学习算法已成功应用于这些领域,例如,网络模型和转录组学已被用于预测化合物的可能靶标。 然而,这方面仍然存在诸多挑战,特别是在缩小药物发开发中生物层次和化学层次的差异。 下面,我们重点介绍下一代机器学习算法将如何在网络生物学的背景下解决这些挑战并加速药物发现和开发过程。
来自药物治疗的多组学数据,以及存储在如dbGAP和GTEx Portal等存储库中的大量基因型数据,为利用机器学习生成综合网络模型带来了所需要的原始生物材料。从机器学习的角度出发,思考如何将这些网络模型与生化上可获得的丰富信息结合起来是及其令人激动的。
多任务学习神经网络(Multi-task-learning neural networks)非常适合这种类型的应用。这类应用的特点是给定的系统中包括横跨多种数据类型(例如,表达谱,化学结构)、包含多种标签(例如对药物,疾病状态的响应)的数据特征。典型的机器学习应用程序定义了一个单一的任务,其中模型被训练以预测单个标签。如果使用相同的输入数据学习新的标签,则新的模型会被训练出来。也就是说,学习任务被视为独立事件。但是,在某些情况下,从一个任务中学习到的重要信息,可以用于另一个任务的学习过程。多任务学习的基本思想是同时共同学习一组任务。单任务学习的目标是优化单个任务的性能,而多任务学习的目标是优化所有任务的性能。多任务学习通过多种表征来学习整个系统,从而一次性学习多项任务。
多任务学习同时学习多个相关任务,并且平衡任务之间的差异和相似性。这种方法基于这样一个前提:学习相关联的概念可以强加学习模型的一般化,提高性能的同时也避免了模型的过度拟合。重要的是,多任务学习神经网络可以整合或合成来自不同来源和分析方法的数据。因此,多任务学习可以同时考虑调控网络的作用关系、来自多组学实验的数据、高通量药物筛选数据、生物活性分析以及药物治疗的表型观察等多个层面来预测给定药物的生理反应及其毒性副作用。

通过将化学实体的结构化数据结合起来,我们有可能利用多任务学习来弥合药物发现的生物和化学方面的差异。例如,可以使用简化的分子输入-行输入系统(Simplified molecular-input line-entry system, SMILES)这种药物表示方法作为模型输入数据。 SMILES表示法将化学物质的结构转换为线性文本串,可以很容易地将其纳入机器学习应用程序中。将SMILES文本串、不同化合物的靶标以及其转录和毒性的相关数据提供给多任务学习算法,可以用来预测新化合物的潜在副作用或可能靶标。此外,我们还可以使用自然语言处理技术,如词嵌入技术,学习基于SMILES的药物的特定属性,这样一来不仅可以对多任务学习进行补充,同时可以帮助我们在随后的药物设计工作中加入或去除某些化合物的关键特性和/或结构特征。
这些机器学习方法也可以用来研究和利用药物化合物的“肮脏”。大多数(如果不是全部)化合物对人体的影响超过其治疗的目的,并且这些影响以剂量和网络依赖的方式变化。多任务学习神经网络非常适合从各种数据类型(例如,不同药物的药代动力学和药效学性质,来自这些药物的细胞筛选的多组学数据等)进行学习,以便更好地理解和预测药物的输入—输出关系(例如,各种化学实体的生物物理和结构性质,它们的分子靶标以及它们诱导的生物学反应)。胶囊网络模型可用于研究复杂疾病,预测出治疗疾病时可能需要抑制的多个靶标。这些预测可以被多任务学习用来识别“肮脏”的化合物,或者对此类化合物进行组合以更好的作用于靶点。因此,我们预见多任务学习与其他深度学习方法结合起来,将有助于解决药物发现中的生物和化学数据整合问题,并创建多层预测网络模型,推进药物的合理化设计。

图1. 深度学习的组织构架在药物开发领域具有很高的应用价值。
微生物研究
人类微生物组包括细菌、古细菌、病毒、真菌、原生动物。这些微生物生活在人体表面或人体内。 每个身体部位的微生物的多样性是惊人的,现在人们认为动态且相互关联的微生物群系统在健康、疾病和发育中起着重要的作用。 人类微生物群的宏基因组数据大量涌现,但将这些数据变得更具有生物学和临床意义仍然是一项重大挑战。 这也为利用下一代机器学习算法力量的网络生物学提供了极好的机会。
不同身体部位的微生物和宿主细胞通过产生、交换和利用小生物分子(主要是代谢物)相互作用。 这些相互作用产生了细胞内、细胞间、物种间和跨生物界的代谢网络。 这为任何给定的微生物群—宿主系统创造了基于共享代谢物产生元代谢网络模型(Meta-metabolic network model)的机会。 这些模型可用于绘制、剖析和理解多种微生物与宿主之间的相互作用,以及预测宿主与其寄生微生物之间可能产生的协同作用和生物质关系(Synergistic and dysbiotic relationship)。
目前为止,我们已经为许多微生物模式生物(例如,大肠杆菌)以及人类细胞构建了代谢网络模型。这些模型提供了代谢物如何通过生物化学反应在给定细胞中相互作用的全局图,我们可以对其进行利用、修改和整合,创建跨越多种生物体或细胞类型的元网络(Meta-networks)。不幸的是,由于数据的稀疏和微生物的数据测定方面的问题,我们对许多微生物中的代谢网络的理解是有限的或根本就不存在。这对于元代谢网络模型的产生提出了重大挑战。这方面的问题也有解决方法,那就是迁移学习(Transefer learning)。
与多任务学习相比,迁移学习旨在从学习不同但相关的任务时获得的知识中进行学习。不同的生物系统具有许多相似特征,这表明在一个系统中生成的数据可以在一定程度上应用于另一个系统。所以这样一来,真正的挑战变成了如何最好地将在给定系统中学到的知识应用于只存在有限数据的新型系统中。
迁移学习可以根据其他系统的观察结果对新系统进行推断和预测。 具体而言,迁移学习使人们能够将用于学习特定任务的模型重新定位为学习不同但相关任务的起点。迁移学习背后的概念很容易适用于生物学中的问题。 以代谢网络为例—生化化合物的不变性(即大肠杆菌中的“葡萄糖”与炭疽杆菌中的“葡萄糖”为相同的有机化合物)为知识的归纳迁移提供了基础。 这意味着在模式生物体中优化的机器学习模型可以重复使用或重新用于在数据稀缺的不同生物体中。
这为研究微生物群落的代谢复杂性开辟了一条令人兴奋的途径,在这里可以“迁移”或使用来自经过模式物种(如大肠杆菌)的代谢网络上的学习信息,应用于未被研究的物种,从而加速我们对微生物群体中多种物种的理解。与迁移学习类似,我们可以概念化一种机器学习模型,这种模型可以利用在较简单系统上获得的知识来理解更复杂的系统。通过这种方式,人们可以建立微生物群与宿主之间的代谢相互作用关系的综合模型。
这些模型可以在横跨健康和疾病状态的生物数据集上进行训练,并用于预测特定物种的消失、引入或生长如何破坏或增强生态系统的代谢平衡,或者此物种是否会产生促进健康的有益代谢物,或损害宿主组织的毒性代谢副产物。值得注意的是,这些先进的机器学习技术和网络生物学方法不一定限于人类健康应用—它们可以很容易地扩展到农业、环境和工业环境中的微生物群研究中。
合成生物学
合成生物学一个重要的研究方向是利用分子元件创建合成基因网络,并利用这些基因线路重编程细胞,赋予它们新的能力。然而,合成基因线路的设计和构建远非直截了当—基因线路的早期版本很少能够按照预期工作,通常需要数周或数月的时间进行反复调谐。基因线路设计的过程主要受到两点的限制:第一是我们对基因线路核心设计原理的理解有限,第二点是缺乏多元化、表征详细的优质元件。如今合成生物学的应用范围扩展到了更大的领域(如健康,农业,能源,环境等),所以我们越来越需要让合成生物学的基因线路设计更加直接和可预测,并且可以进一步提高时间效率。这为深度学习方法创造了绝佳机会,我们将在下面重点介绍。
合成生物学基因线路存在多个调控层次。在基础水平,线路中存在单独的分子组分,例如基因,启动子,操纵子,终止子和核糖体结合位点。 在中间水平,存在由多个组分组成的调控单元,例如基因—启动子对。 在高级水平,调控单元之间通过相互作用产生特定的基因线路,例如,两个基因—启动子对可以排列在相互抑制的网络中产生双稳态开关。在每一个层次,我们都可以用序列代表来定义某一水平的调控方式、组成性关系(例如,空间和方向的排列)、以及影响功能的生物分子、分子组分和/或子组分之间的相互作用。
我们可以生成、测序和功能表征大量且多样化的分子元件,调节单元以及合成基因线路,为深度学习方法创建适当的训练数据集。 功能表征可以包括量化RBS的强度、启动子—基因对的Hill系数、以及基因线路的响应时间等等。 由于深度学习方法在很大程度上依赖于大量数据,因此我们可以将机器与基于板的分析(自动化)结合到实验过程中,开发和实施快速的实验工作流程,对元件、调控单元以及基因线路进行表征。
我们可以设想使用测序和功能表征数据来产生多种生物调控层次的基因线路预测模型。为此,可以开发一个多阶段的深度学习模型,这种模型可以从嵌入有生物序列的学习模型到嵌入有调控模体和线路构建的学习模型中学习每一种基因线路组织方式的的本质。例如,可以利用递归神经网络来编码不同元件的序列,其中序列可以被视为特定的“句子”,其允许模型学习特定的“句子属性” 包括样式,语法和主题,这些序列等同于元件本身的序列信息(启动子,结合区和终止子的DNA序列)。
此外,卷积神经网络可用于编码调控单位和合成基因线路拓扑结构上的特征。我们可以训练算法学习不同元件的序列—功能关系,以及调控单位和合成基因线路的组成—功能关系。利用这种方式,该模型可以从调控(网络控制)角度和拓扑(网络架构)角度学习合成基因线路的关键性质。
生成的深度学习模型可用于分析合成生物学的基本设计原则。相应地,也可用于产生性能增强或功能新颖的元件(例如,诱导型启动子,操纵基因等),从而提高可用于合成生物学设计工作的分子元件的数量和多样性。深度学习模型还可用于设计和识别新的调控单元和合成基因网络。例如,对于给定的期望功能,该模型可用于生成一组产生所述功能的基因线路。我们将这种方法与数学模型相结合,可以非常迅速地对数千个潜在的线路进行迭代。其中最有价值的候选线路可以进行合成,测试和进一步验证。这方面的进步将有助于合成生物学的快速设计,促进复杂的合成基因线路在生物医学领域的广泛应用。
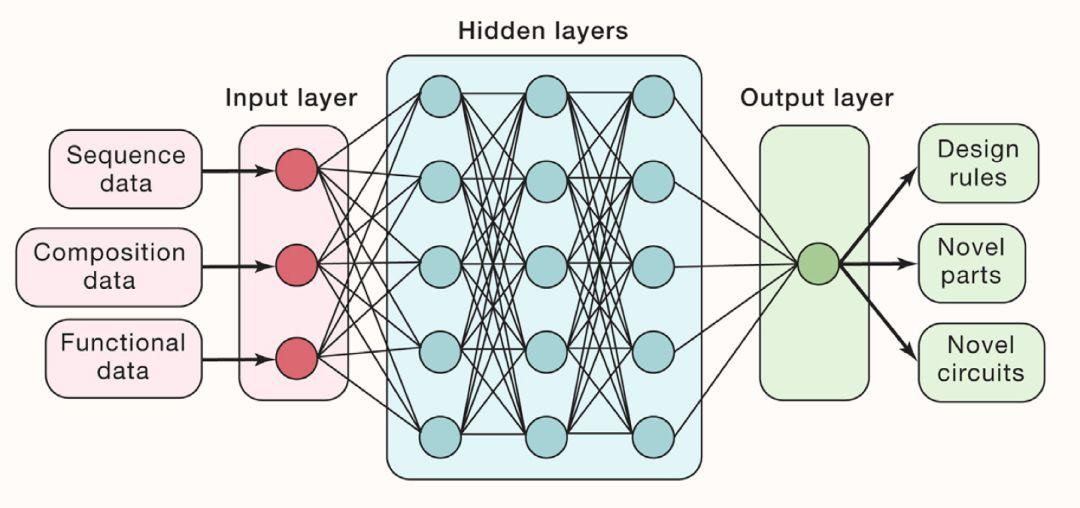
图2. 合成生物学的深度学习应用包括新型设计规则、分子元件以及基因线路的预测。
挑战与展望
从上面的讨论中我们可以清楚地看出,网络生物学与下一代机器学习的交叉研究拥有着巨大的机遇。 但是,还有很多艰难的挑战需要我们客服。 其中最关键也最重要的是大型数据集。 深度学习方法和其他下一代机器学习方法对于数据是及其饥渴的。 我们生活在生物学和医学的大数据时代,我们在生物的不同层次收集大量的数据集。 尽管从生物系统获取的数据可能非常复杂,反应生物系统的不同方面的变量可能包含有数千个,但是,大多数生物数据集的数量级仍然太小,无法深度学习算法的要求。
当然,我们有很多选择来应对这一挑战。首先是收集大型且注释良好的数据集。多组学数据集可能过于昂贵,因此我们可以考虑替代方案来补充或补足这些数据。由于许多深度学习算法已经成功地应用于诊断背景下的成像数据,所以增加利用成像数据(包括视频)来表征细胞的形态或表型变化(例如响应药物治疗)将会一种不错的选择。我们可以通过建立具有荧光或比色读数的细胞系来来表征各种治疗方法或环境扰动下的细胞反应。许多生物数据集小而稀疏的特性也给机器学习研究人员带来了一个有趣的挑战—即专门设计来处理此类数据集的新一代深度学习算法。
另一种可能的选择是生成具有实际数据属性的计算机数据。对于深度学习背景下的图像分析,研究人员通常使用生成对抗网络(Generative adversarial networks, GAN)来完成,该方法可以创建与训练数据类似的数据集。 GAN是深度神经网络架构,由两个互相对立的神经网络组成—一个是生成模型(Generative model),用于产生与训练数据集分布相似的新数据,另一个是区分模型(Discriminative model),即对手,用于评估新数据并确定它是否属于实际的训练数据集。
两个神经网络之间的竞争会逐渐的改进算法,直到生成的数据集与训练数据集无法区分为止。这种机器学习方法可以很容易地在网络生物学的多组学数据集找到应用的价值。一个简单的例子就是使用GAN生成更大的基因表达数据集,这些数据集将有助于生成转录调控的预测模型。
大多数下一代机器学习模型的“黑箱”性质为生物应用带来了新的挑战。从生物学的角度来解释给定模型的输出通常是非常困难的,从而限制了该模型在生物机制和网络架构分析中的效果。当然,情况并非总是如此,特别是对于更简单的机器学习方法。例如,稀疏线性回归模型(Sparse linear regression model,比如elastic net, lasso and ridge regression)可以用来学习每个特征相对权重的最佳系数。在这种情况下,模型系数可以告知研究人员模型中每个特征的相对“重要性”。然而,对于更先进的机器学习方法,例如深度神经网络,训练过程对输入数据的处理方式使得我们难以确定特征的相对重要性或特征是否与输出结果存在正相关或负相关。我们迫切需要开发出一种手段,将深度学习的“黑箱”转变为在生物学角度富有意义且可解释的“白箱”。
在揭示和利用生物复杂网络的方向我们还有很长的路要走 ,机器学习本身还远未实现其在生物研究领域的潜力。 尽管如此,利用机器学习更好的理解复杂生物网络的领域的发展让网络生物学研究可以拥有一个令人激动的未来。
参考文献
翻译:孟凡康
编辑:王怡蔺
推荐阅读
2018集智俱乐部年会报名
点击图片了解年会详情
2018年12月31日,集智俱乐部(Swarma Club)将联合多家科技、学术组织,在北京举办一年一度的学术晚会。本届晚会的主题是“奇点临近”(The Singularity is Near)。
在这样一个特殊的Party之中,你可能邂逅正在思索深度学习与量子纠缠的“青椒”,也可能偶遇正在实践颠覆式创新的创业者,还可能与正在寻找投资机会的金主撞个满怀,更有机会和某个行业大咖侃侃而谈。在这里,找到与您志同道合的伙伴。
这里,是平等,也是跨界。

集智俱乐部QQ群|877391004 商务合作及投稿转载|swarma@swarma.org 搜索公众号:集智俱乐部 加入“没有围墙的研究所” 让苹果砸得更猛烈些吧!
◆ ◆ ◆

原文始发于微信公众号(集智俱乐部):集智











