Nat. Mach. Intell. 速递:可解释双线性注意力网络提高药物靶点预测能力

关键词:深度学习,药物发现,蛋白质预测,分布外泛化,可解释机器学习


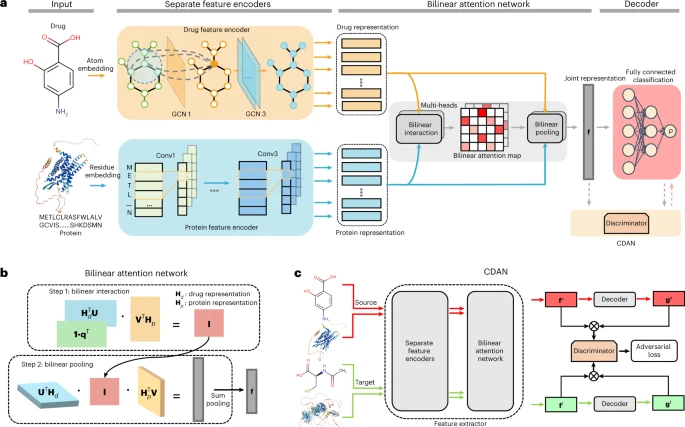
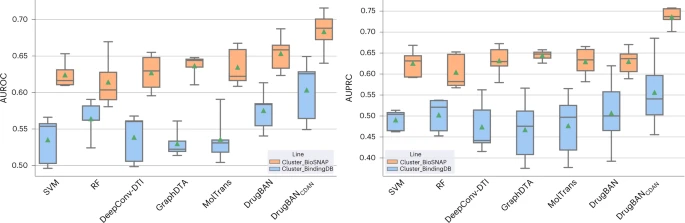
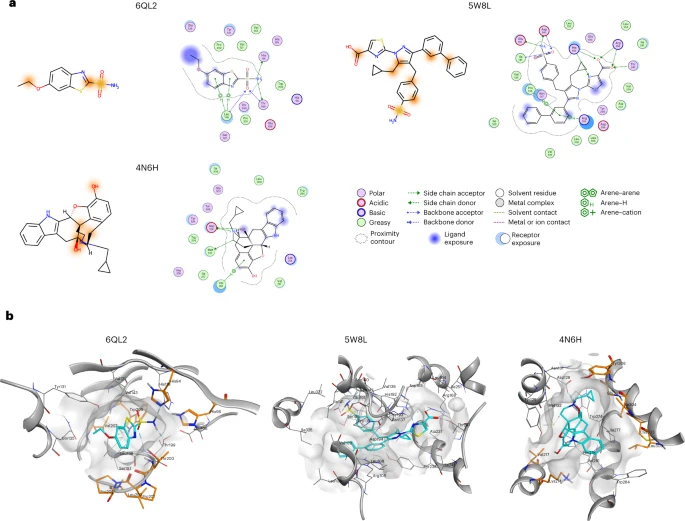
论文题目:Interpretable bilinear attention network with domain adaptation improves drug–target prediction 论文来源:Nature Machine Intelligence 论文链接:https://www.nature.com/articles/s42256-022-00605-1
复杂科学最新论文

推荐阅读
-
研究速递:GNN能否学习更好的分子表征用于药物发现? -
老药新用治疗新冠疾病?网络医学框架加速药物研发 -
Sci. Adv. 综述:机器学习方法在社会和健康科学中的应用 -
《张江·复杂科学前沿27讲》完整上线! -
成为集智VIP,解锁全站课程/读书会 -
加入集智,一起复杂!










