整合生命组学数据,揭示生命复杂系统构成原理
导语
谢林海 | 作者
转录组学
蛋白质组学
单细胞转录组学
阅读材料较长,为了更好的阅读体验,建议您前往集智斑图沉浸式阅读,并可收藏感兴趣的论文。

斑图地址:https://pattern.swarma.org/articles
转录组学
转录组学
转录组学(Transcriptomics)是研究细胞、组织或生物体中所有转录产物(即RNA分子)的一门科学。转录组包括信使RNA(mRNA)、非编码RNA(如小核RNA(snRNA)、小核仁RNA(snoRNA)、长链非编码RNA(lncRNA)以及微RNA(miRNA)等)。转录组学的主要目的是全面分析和解释基因表达及其调控机制。
-
转录组:指一个特定时间点、特定条件下,某一细胞、组织或生物体中所有的RNA转录产物的集合。 -
转录产物:主要包括mRNA和各种类型的非编码RNA,如rRNA、tRNA、miRNA、lncRNA等。 -
转录组数据通常被组织成一个二维矩阵,每一行代表一个基因,而每一列代表一个样本,表示每个基因在不同样本中的表达水平,可以反映出其在不同条件下的活跃程度。样本可以来自不同个体的生物样本,不同实验条件下的样本,或者不同时间点的样本等。
缺点:只能检测已知序列,灵敏度和动态范围有限。
缺点:成本较高,数据处理复杂。
-
基因表达谱分析:通过比较不同条件下的基因表达差异,识别与特定生物学过程或疾病相关的基因。应用于癌症、神经退行性疾病、感染性疾病等研究中。 -
基因调控网络构建:研究基因表达的调控机制,构建基因调控网络,揭示基因间的相互作用和调控关系。有助于理解复杂的生物学过程,如发育、细胞分化、应激反应等。 -
新转录本的发现:RNA-Seq技术可以发现新的转录本和可变剪接事件,揭示基因表达的复杂性和多样性。帮助完善基因组注释,拓展对基因功能的认识。 -
非编码RNA的研究:非编码RNA在基因调控中起重要作用,如miRNA参与基因沉默,lncRNA参与染色质修饰和转录调控。研究非编码RNA的功能和机制,揭示其在发育和疾病中的作用。
例子:对实验组和对照组的基因表达做差异分析,找出相对于对照组中,实验组的上下调基因:
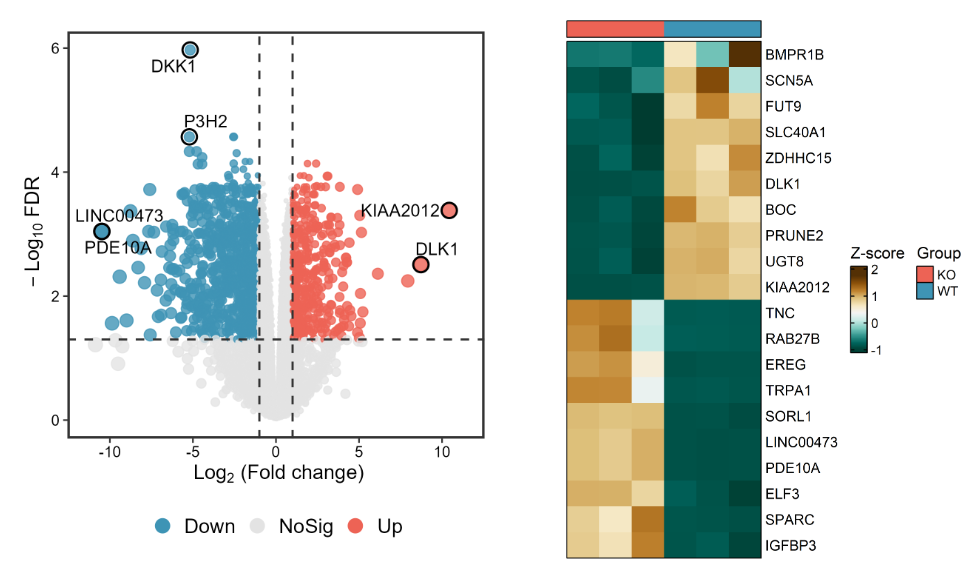
2. 功能富集分析(Functional Enrichment Analysis):对差异表达基因进行基因本体(GO)富集分析、通路分析(如KEGG)、GSEA(基因集富集分析)等。
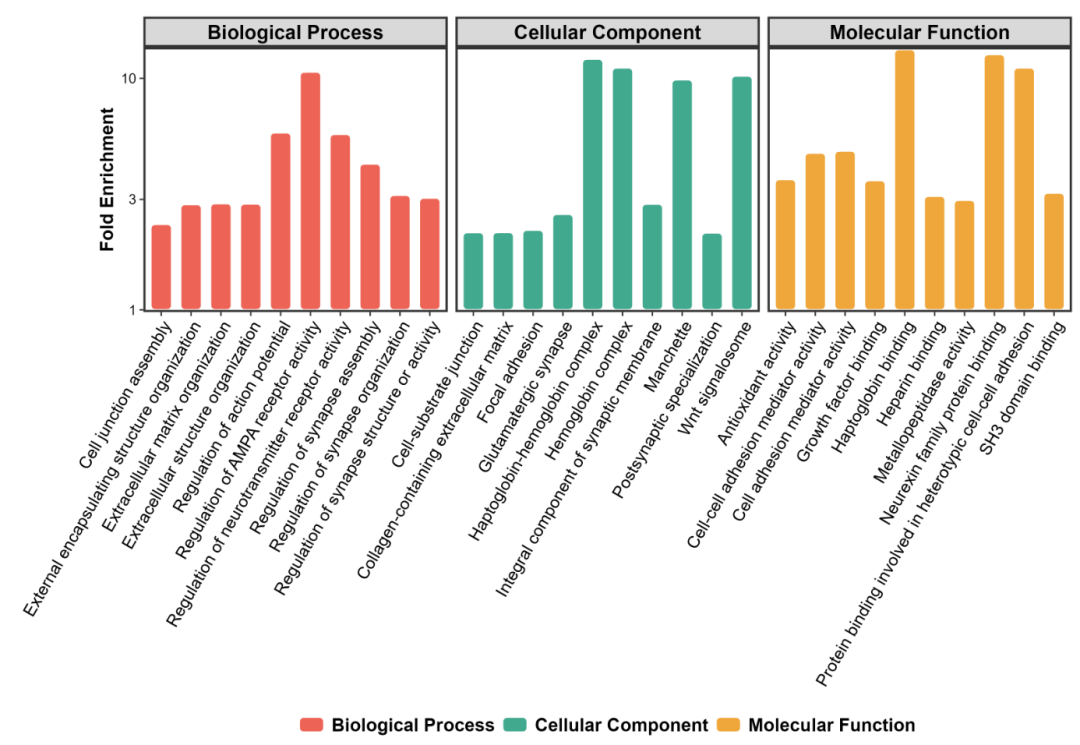

例子:通过对动态切割聚类,找到稳健的基因模块,并将模块的特征基因和临床性状做关联,以此来找到和目标临床形状关联的基因模块。
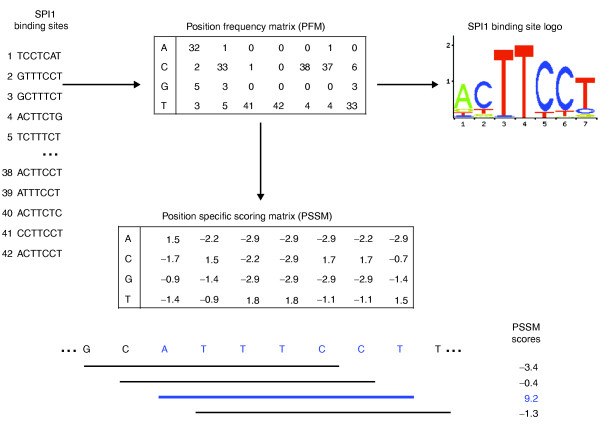
例子:通过统计每个碱基在不同位置的频率(PFM),转换为评分矩阵(PSSM),并使用这些评分矩阵计算新的DNA序列与SPI1结合位点的匹配分数。这一过程帮助识别哪些DNA序列可能是转录因子的结合位点。
5. 可变剪接分析(Alternative Splicing Analysis):通过软件如rMATS, MISO分析不同条件下的可变剪接事件。
蛋白质组学
蛋白质组学
蛋白质组学(Proteomics)是研究细胞、组织或生物体内所有蛋白质的结构与功能的一门科学。蛋白质是生命活动的直接执行者,研究蛋白质的表达、修饰、相互作用及其在生物过程中的作用,对于理解生命现象和疾病机制具有重要意义。以下是蛋白质组学的详细介绍:
一、蛋白质组学的基本概念与数据描述
指在特定时间、特定条件下,某一细胞、组织或生物体中所有蛋白质的集合。旨在全面分析蛋白质的表达水平、修饰状态、相互作用和功能。
蛋白质组数据通常也被组织成一个二维矩阵,每一行代表一个蛋白质,每一列代表一个样本,表示每个蛋白质在不同样本中的丰度,可以反映其在不同条件下的功能和活性。样本可以是来自不同个体的生物样本,不同实验条件下的样本,或者不同时间点的样本等。
二、蛋白质组学的研究方法
双向电泳(2-DE):通过等电聚焦(IEF)和SDS-PAGE分离蛋白质,根据蛋白质的等电点和分子量进行分析。
优点:分辨率高,适用于复杂样品。
缺点:操作复杂,无法检测低丰度或难溶的蛋白质。
质谱分析(Mass Spectrometry, MS):质谱技术通过离子化蛋白质或肽段,测量其质荷比(m/z),用于蛋白质鉴定和定量。
优点:灵敏度高,适用于高通量分析。
缺点:需要复杂的数据分析和解释。
蛋白质芯片(Protein Microarray):利用固定在芯片上的特异性抗体或配体,检测样品中相应蛋白质的表达水平。
优点:高通量,适用于蛋白质表达谱分析。
缺点:依赖于已知蛋白质和抗体的质量。
-
蛋白质表达谱分析:通过比较不同条件下的蛋白质表达差异,识别与特定生物学过程或疾病相关的蛋白质。 -
蛋白质修饰分析:研究蛋白质的翻译后修饰(如磷酸化、乙酰化、糖基化等),揭示其在信号传导和功能调控中的作用。 -
蛋白质相互作用研究:通过蛋白质互作网络,共免疫沉淀(Co-IP)、酵母双杂交(Y2H)、拉曼光谱等技术,研究蛋白质间的相互作用网络。有助于理解蛋白质在细胞内的功能网络和调控机制。 -
生物标志物发现:通过蛋白质组学研究,寻找疾病特异性蛋白质作为生物标志物,用于早期诊断和预后评估。例如,癌症中的特异性蛋白质标志物。 -
药物靶点鉴定:通过蛋白质组学研究,鉴定与疾病相关的蛋白质作为潜在的药物靶点,推动新药开发。例如,针对特定蛋白质的抑制剂开发。
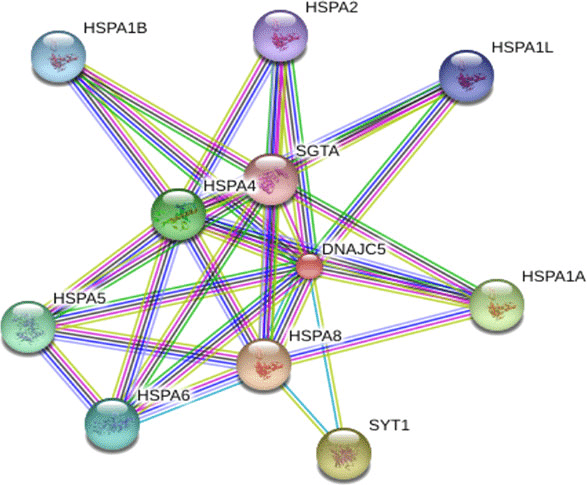
例如:用DNAJC5查询STRING蛋白-蛋白相互作用网络。蛋白质之间的彩色线表示各种类型的相互作用证据。
4. 翻译后修饰分析(Post-translational Modification Analysis):识别并分析蛋白质的翻译后修饰(如磷酸化、乙酰化、泛素化),研究其功能意义。
单细胞转录组学
单细胞转录组学
单细胞转录组学(Single-cell Transcriptomics)是研究单个细胞中基因表达的技术,旨在揭示细胞间的异质性和个体细胞在不同生物过程中的特定角色。通过对单细胞的RNA进行测序,单细胞转录组学能够提供高分辨率的基因表达谱,揭示组织和器官在分子水平上的复杂性。以下是单细胞转录组学的详细介绍:
一、单细胞转录组学的基本概念与数据描述
指在单个细胞水平上,某一特定时间点和条件下的所有RNA转录产物的集合。通过分析单个细胞中的基因表达,研究细胞异质性、细胞命运决定及其在生物学过程中的功能。
单细胞转录组数据通常也被组织成一个二维矩阵,每一行代表一个基因,每一列代表一个单细胞,表示每个基因在不同细胞中的表达水平,可以反映其在单细胞层次上的活跃程度和功能状态。
-
荧光激活细胞分选(FACS):利用荧光标记和流式细胞术分选单个细胞。 -
微流控芯片技术:通过微流控装置捕获和分离单个细胞。 -
激光捕获显微切割(LCM):利用激光显微切割技术从组织切片中分离单个细胞。
-
Smart-seq:扩增单细胞RNA,适用于全长转录本测序。 -
Drop-seq:通过微滴技术将单个细胞和微珠包裹在一起,进行高通量测序。 -
10x Genomics Chromium:采用微流控平台,实现高通量单细胞测序。
-
细胞异质性分析:通过解析单细胞水平的基因表达,揭示组织中不同细胞类型的异质性。 -
细胞谱系追踪:研究细胞在发育和分化过程中的动态变化,追踪细胞谱系关系。在发育生物学、再生医学等领域具有重要应用。 -
疾病研究:通过单细胞分析,揭示疾病组织中的细胞异质性和特异性细胞类型。帮助理解疾病的发生和发展机制,如癌症中的肿瘤微环境、免疫逃逸机制等。 -
药物反应和耐药机制研究:研究单个细胞对药物的反应,揭示耐药细胞的特征和机制。为个性化治疗和新药开发提供依据。
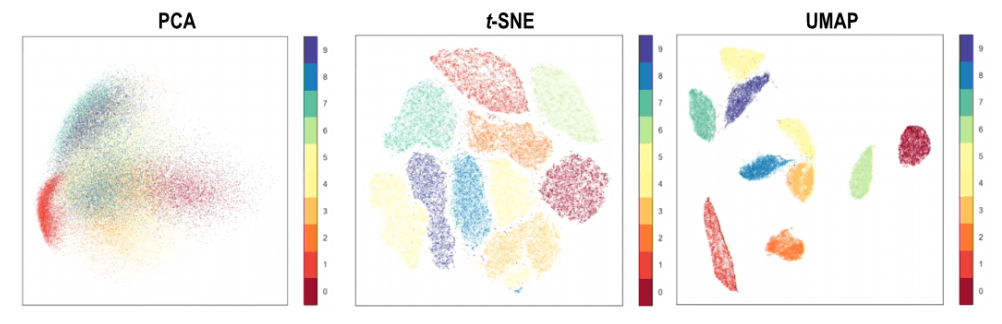
例如:通过PCA、tSNE和UMAP方式对细胞进行降维分析
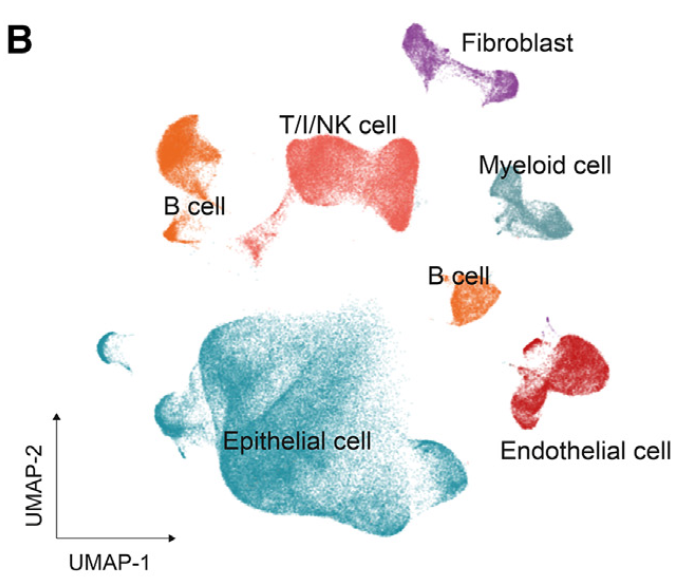
例如:通过经典的细胞分子标志物来注释结直肠癌的细胞亚群
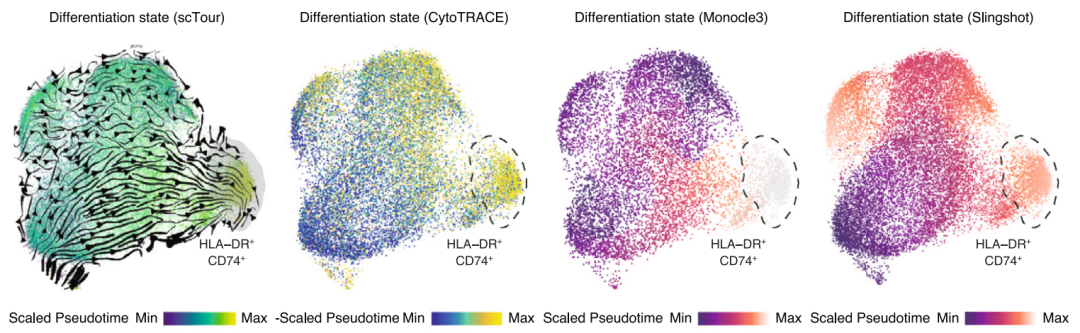
例如:通过4种伪时序分析算法定位到中性粒细胞的终末分化亚群
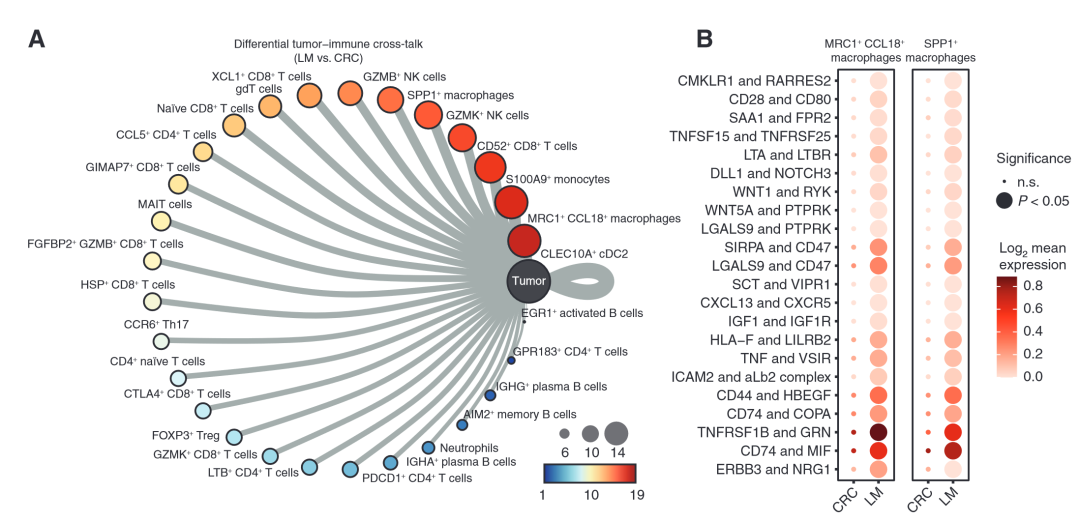
例如:基于CellChat研究肿瘤细胞和其它细胞的细胞通讯强度
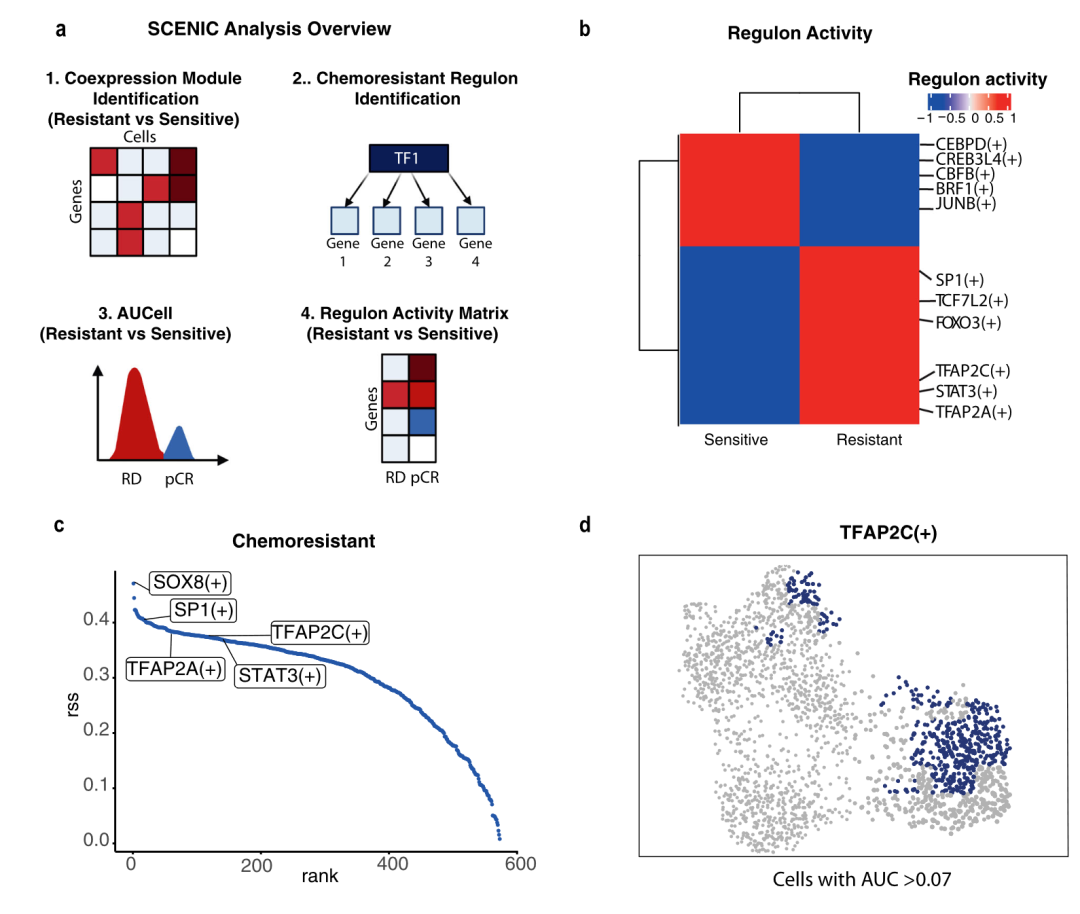
例如:基于SCENIC来分析耐药细胞株的驱动转录因子
10. 细胞谱系树构建(Cell Lineage Tree Construction):使用工具如TSCAN, FateID构建细胞谱系树,研究细胞分化路径和谱系关系。
空间转录组学
空间转录组学
空间转录组学(Spatial Transcriptomics)是一种结合空间信息和转录组学数据的技术,旨在揭示组织中基因表达的空间分布。通过在组织切片上保持细胞空间位置的同时,进行RNA测序,空间转录组学可以提供组织结构和功能的高分辨率图谱。以下是空间转录组学的详细介绍:
一、空间转录组学的基本概念与数据描述
空间转录组:指在保留细胞空间位置信息的情况下,对特定组织或器官中所有RNA转录产物的集合。通过结合空间信息和RNA测序,研究组织中基因表达的空间分布及其在生物学过程中的作用。
空间转录组数据经常被组织成一个二维矩阵,每一行代表一个基因,每一列代表一个空间位置或区域,表示每个基因在不同空间位置的表达水平可以反映其在特定组织结构中的活跃程度和功能状态。这些位置可以是组织切片上的不同点,通过技术如空间转录组学测序或其他空间分辨的测序方法获得。位置信息通常以坐标形式(如 x 和 y 坐标)存储,表示在组织切片上的具体位置。
优点:具有较高的空间分辨率(约55微米),能够生成组织的高质量空间表达图谱,适用于各种组织类型的研究。
优点:极高的空间分辨率(2微米),能够解析细胞水平的基因表达异质性,适用于研究复杂组织的微观结构。
优点:纳米级空间分辨率(约500纳米),能够详细解析细胞内结构和基因表达,适用于高精度空间转录组分析。
-
组织结构与功能分析:通过解析组织中基因表达的空间分布,揭示不同细胞类型和区域的功能差异。比如:研究癌症组织中肿瘤细胞、免疫细胞和基质细胞的空间分布及其相互作用,帮助理解肿瘤微环境的复杂性和异质性。 -
微环境研究:研究组织中不同细胞类型及其微环境的相互作用,揭示细胞间的通信和调控机制。比如:在神经科学中,研究大脑中神经元和胶质细胞的空间分布和功能连接,理解神经网络的结构和功能。 -
疾病机制研究:通过空间转录组学分析,揭示疾病组织中的特定细胞类型和区域的基因表达变化。比如:在心血管疾病研究中,分析心肌梗死区域内不同细胞的空间基因表达,揭示心肌修复和重塑过程中的关键基因和路径。 -
发育与再生研究:研究胚胎发育过程中基因表达的空间动态变化,揭示发育调控机制。比如:在发育生物学中,研究胚胎不同发育阶段的基因表达变化,理解器官发育和细胞分化的空间调控。 -
药物反应和耐药机制研究:分析药物处理前后组织中基因表达的空间变化,揭示耐药细胞的特征和机制。比如:在肿瘤学中,研究抗癌药物处理后的肿瘤细胞基因表达变化,寻找耐药细胞及其空间分布,为个性化治疗提供依据。
-
生物标志物发现:通过空间转录组学研究,寻找疾病特异性基因表达模式和空间分布特征,作为潜在的生物标志物。比如:在肝病研究中,发现肝纤维化区域内特异性表达的基因,作为诊断和预后的生物标志物。
-
神经网络研究:研究大脑中神经元和其他细胞类型的空间基因表达,揭示神经网络的功能连接和调控机制。比如:在阿尔茨海默病研究中,分析大脑不同区域的基因表达,理解疾病过程中神经元和胶质细胞的变化和相互作用。
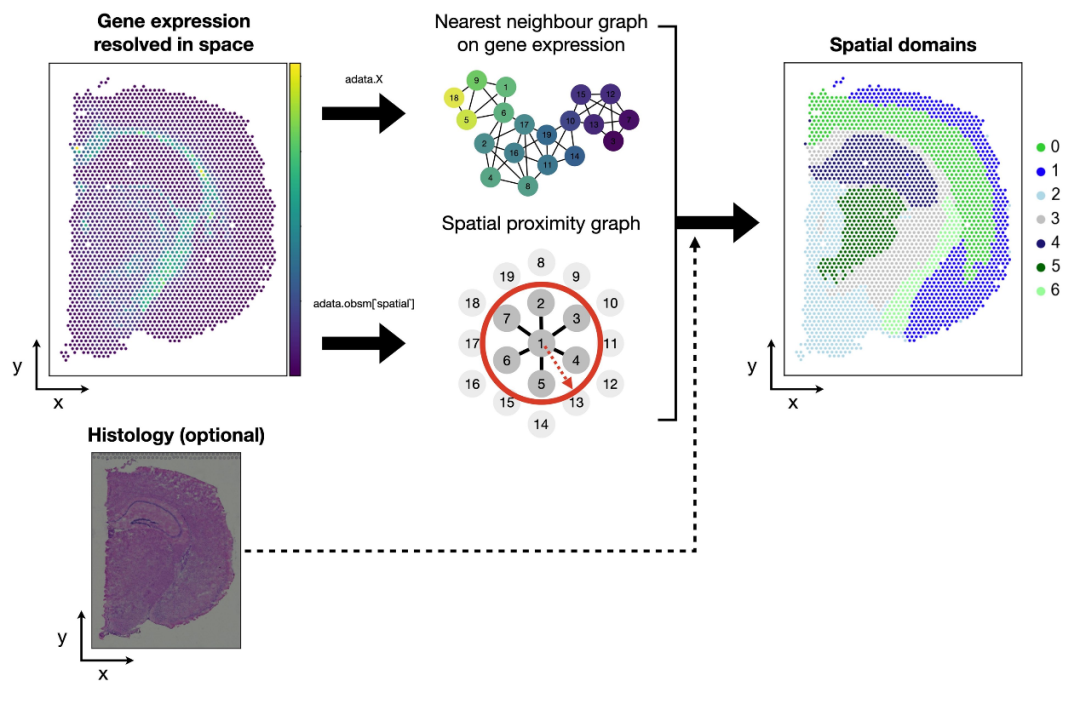
1. 使用降维方法(如PCA, t-SNE, UMAP)和聚类算法(如Louvain, K-means)进行空间点聚类,识别不同空间区域的基因表达模式。
2. 空间可视化(Spatial Visualization):将基因表达数据投影到组织切片图像上,生成空间基因表达热图和空间点图(如Seurat, Scanpy中的空间模块)。
例如:在HD空间转录组数据中可视化FCGBP的表达,该分子标志物是杯状细胞的marker
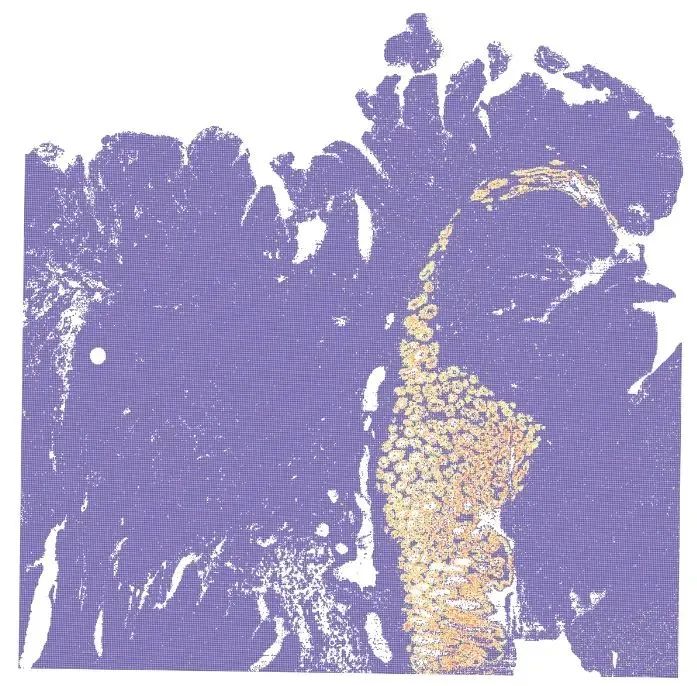
例如:基于Cell2Location反卷积空间Spot的细胞组成

例如:查看空转中每个Spot的通路活性评分,以此来分析空间中的通路激活状态。
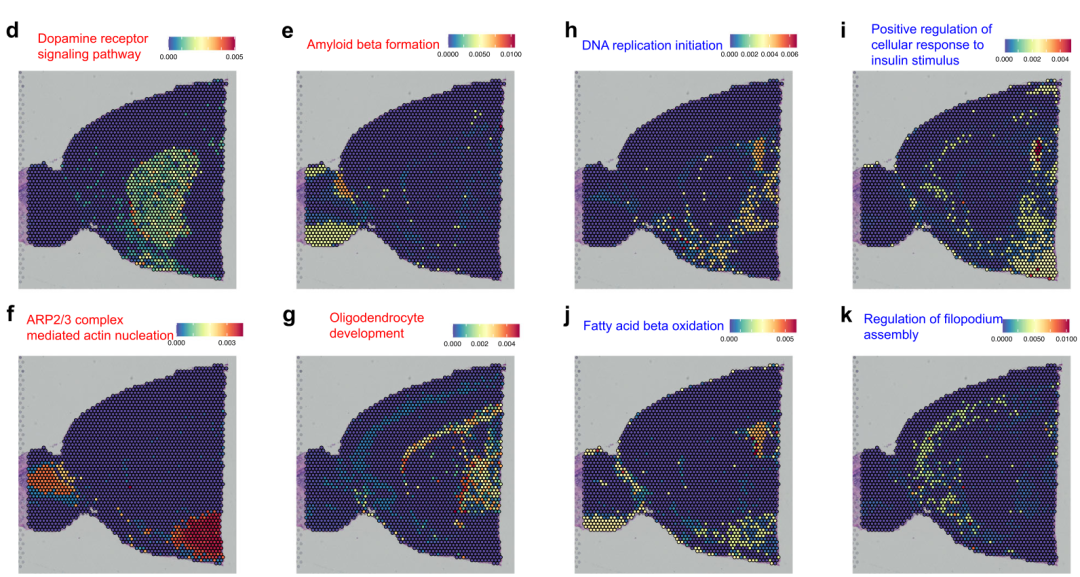
10. 空间域差异分析(Spatial Domain Differential Analysis):比较不同空间域的基因表达和功能特征,研究空间域之间的差异。
例如:基于CellChatV2来计算不同亚群在SPP1信号中的交互强度

生命复杂性读书会招募中
集智俱乐部联合西湖大学理学院及交叉科学中心讲席教授汤雷翰,国家蛋白质科学中心(北京)副研究员常乘、李杨,香港浸会大学助理教授唐乾元,北京大学前沿交叉学科研究院研究员林一瀚,中国科学院分子细胞科学卓越创新中心博士后唐诗婕,共同发起「生命复杂性:生命复杂系统的构成原理」读书会,从微观细胞尺度、介观组织器官尺度,到宏观人体尺度,梳理生命科学领域中的重要问题以及重要数据,希望促进统计物理、机器学习方法研究者和组学研究者之间的深度交流,建立跨学科合作关系,激发新的研究思路和合作项目。读书会从2024年8月6日开始,每周二晚19:00-21:00进行,持续时间预计10-12周。欢迎对这个生命科学、物理学、复杂科学、计算机科学深度交叉的前沿领域感兴趣的朋友加入!











