从2021年AlphaFold 2成功预测2亿种蛋白质结构开始,生命科学领域的人工智能研究进入了一个前所未有的快速发展期。在短短三年间,从DNA到RNA,从分子到细胞,一系列突破性的生命语言大模型相继问世。这些模型不同于处理文本和图像的传统AI,它们专门解析生命的多层次语言:AlphaFold 3能以接近实验精度预测生物分子复杂结构;RhoFold/RhoDesign实现RNA的精确分析与设计;基因表达预测系统GET可以模拟5000多种人类细胞类型的转录调控。更具突破性的是,斯坦福大学开发的多智能体虚拟实验室,通过集成多个AI模型,实现了从分子设计到实验验证的全流程自动化。随着全球细胞图谱计划的推进和AI虚拟细胞的构想,我们正在见证生物学研究从观察科学向工程科学的历史性转变,尽管生命系统的终极复杂性仍然远超我们的认知范畴。
论文题目:Learning the language of life with AI
论文地址:https://www.science.org/doi/abs/10.1126/science.adv4414?af=R
2021: AlphaFold 2解决蛋白质折叠难题
2021年,在ChatGPT引发生成式人工智能(AI)热潮前一年,AlphaFold 2[1]破解了长达50年的蛋白质折叠难题,实现了通过氨基酸序列预测超过2亿种蛋白质三维(3D)结构。这一突破开启了生命科学领域大语言模型(Large Language Models,LLMs) [2]的空前发展,而这仅是开端。
最近几个月,我们迈入了基础模型的超速发展阶段。这些模型通过海量数据集上进行预训练,能够执行多种任务,帮助我们理解蛋白质、RNA、DNA和配体的结构、生物学特性、进化过程及设计方式,同时揭示它们之间的生物分子相互作用。与处理文本、音频和图像的多模态大语言模型(如GPT-4、Gemini和Claude)不同,这些生命大语言模型(Large Language of Life Models,LLLMs)具有多组学(multi-omics)特性,不仅表现为多模态性,还涉及分子生物学的多个层次。例如,Evo[3]是一个基础模型,它在270万个多样化的噬菌体和原核生物基因组(prokaryotic genomes)上进行了训练后(相当于约3000亿个DNA碱基),可以预测DNA、RNA或蛋白质中变异对结构和功能的影响,评估这些基因对细胞功能的重要性,并能够生成新的DNA序列。
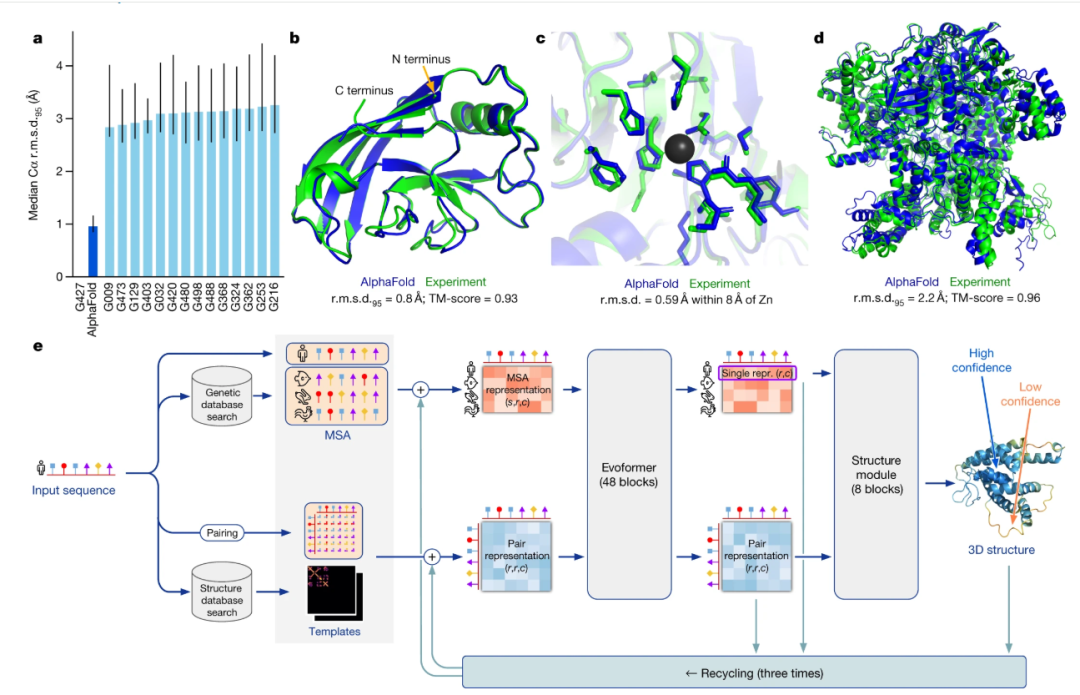
用AlphaFold3预测的精准蛋白质结构 | 来源:Abramson, J., Adler, J., Dunger, J. et al. Accurate structure prediction of biomolecular interactions with AlphaFold 3. Nature 630, 493-500 (2024).
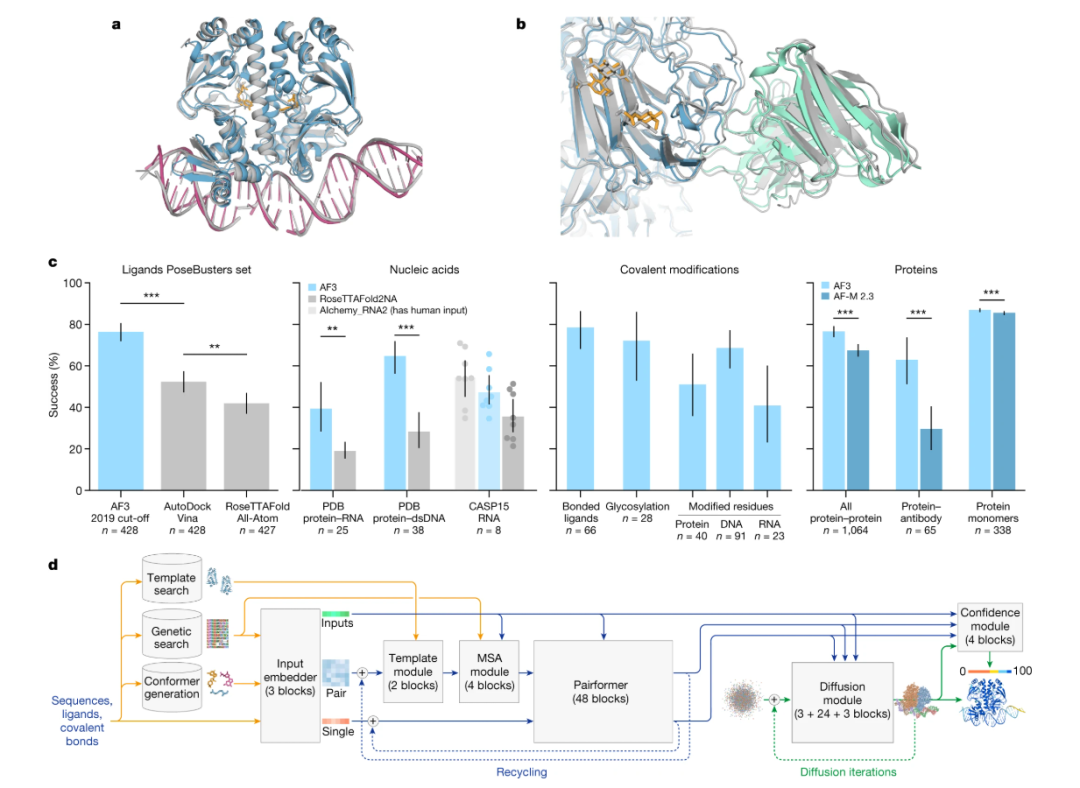
为展现该领域突飞猛进的发展速度,让我们详细介绍近期报道的模型:AlphaFold 3[4] 能预测由蛋白质、DNA、RNA、小分子和配体组合形成的复合物三维结构,其预测精度达实验级水平——80%的蛋白质-配体复合体预测结果与实验误差在2 Å(注意1 Å等于0.1纳米,或十亿分之一米)。同样,Boltz-1[5] 在预测三维生物分子互作方面达到了与AlphaFold 3相当的精度,且具有完全开源的优势。MassiveFold[6] 作为AlphaFold的优化版本,实现了大规模并行计算,显著缩短了计算时间。EVOLVEpro[7]是一个面向AI辅助蛋白质工程的蛋白质语言模型,而PocketGen[8]则可以定义蛋白质-配体互作的原子结构。其他重要模型包括:用于深入理解蛋白质在健康与疾病状态下互作机制的PIONEER[9],以及用于高效设计新型抗体的抗体诱变增强处理系统[10](Antibody Mutagenesis-Augmented Processing,AbMAP)。后者已成功设计出对新型冠状病毒(SARS-CoV-2)具有20倍以上结合亲和力的抗体。
AF3 准确预测生物分子复合物的结构。来源:Abramson, J., Adler, J., Dunger, J. et al. Accurate structure prediction of biomolecular interactions with AlphaFold 3. Nature 630, 493–500 (2024). https://doi.org/10.1038/s41586-024-07487-w
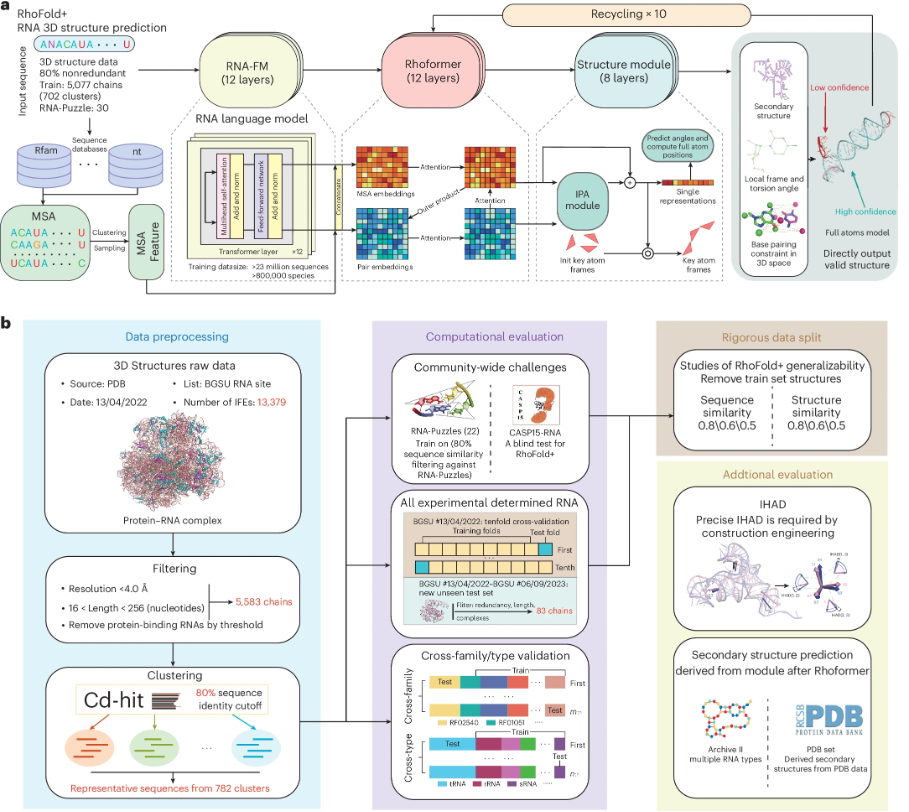
除了蛋白质,RhoFold也值得关注[11]。RhoFold被训练用于从核酸序列预测三维RNA构象。由同一团队研发的RhoDesign[12]则用于RNA适配体的设计(RNA适配体是指能够以高亲和力结合目标蛋白的小型RNA分子)。针对细胞类型特异性的转录现象,通用表达转换器(General Expression Transformer, GET)[13]可以准确预测各类人体细胞中的RNA转录模式。最新的DNA语言模型[14] 能够评估人类基因组编码区和非编码区变异的功能效应,已完成约90亿个可能的单核苷酸变异位点分析。甲基化大语言模型MethylGPT[15]与CpGPT[16](二者研究尚处预印本阶段)致力于表观遗传分析,如生物学年龄推算。SyntheMol[17]则助力新型抗生素的设计和验证,展示了如何从约300亿种化合物中筛选出针对不动杆菌(Acinetobacter baumannii)的抗生素,并完成实验验证。
在细胞层面,存在单细胞相似性(Single-Cell Similarity, SCimilarity)[18]。这种方法利用近似最近邻搜索来识别细胞类型。它是基于相似性的分类,采用了机器学习技术。虽然我们曾经认为人体内大约有200种细胞类型,但现在这个数字被认为超过5000。SCimilarity模型及其同类技术的突破源于“人类细胞图谱”全球计划的推进——来自100个国家的3000名科学家已完成6200万单细胞图谱绘制,正向十亿级细胞数据库迈进。
RhoFold+ 的架构和用于性能评估的任务。来源:RhoFold+ Shen, T., Hu, Z., Sun, S. et al. Accurate RNA 3D structure prediction using a language model-based deep learning approach. Nat Methods 21, 2287–2298 (2024). https://doi.org/10.1038/s41592-024-02487-0
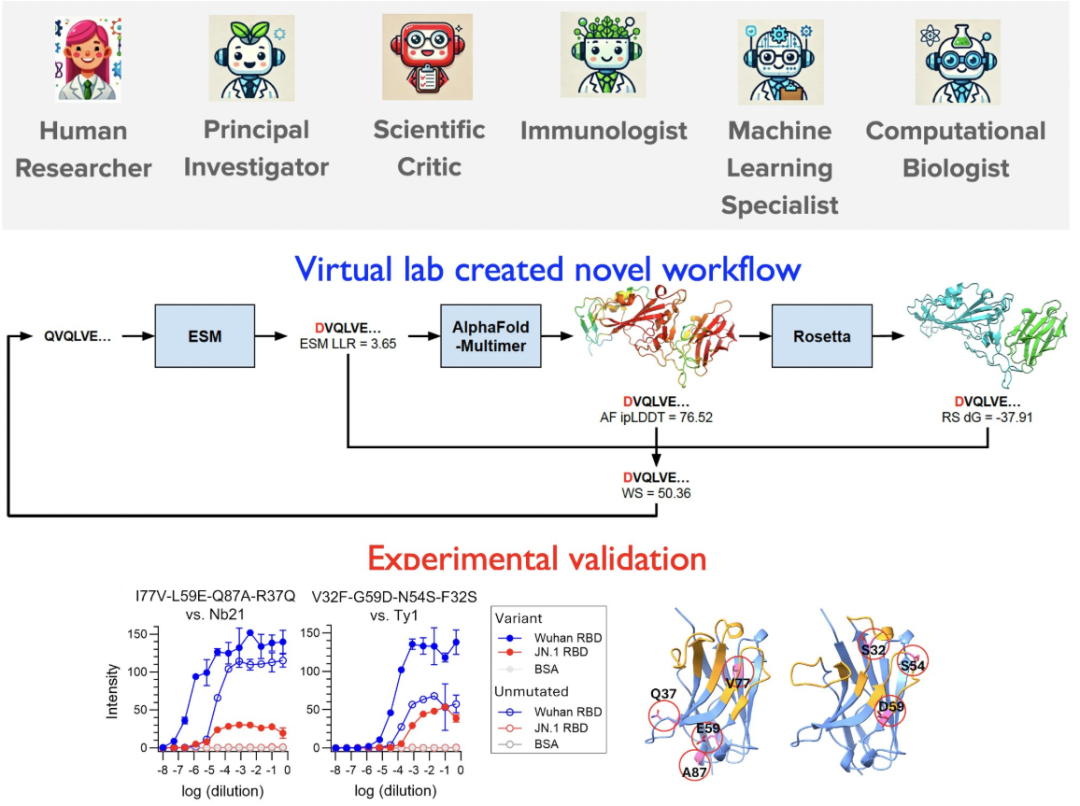
在单一语言模型取得长足进展之际,我们正迈入多智能体协同的计算机辅助科学发现的时代。斯坦福大学James Zou团队开发的“虚拟实验室”(Virtual Lab)系统就是典型案例——该系统集成五位不同领域专家型智能体(首席研究员、免疫学家、机器学习专家、计算生物学家及科学评论员),通过融合AlphaFold-Multimer、Rosetta与进化尺度建模(ESM)三大语言模型,实现了针对SARS-CoV-2纳米抗体的自动化设计。在最小化人工干预的前提下,五位智能体通过定期自主会商,快速研发出两种高效纳米抗体(一种工程化微型抗体片段),其效果已通过实验室检测验证。
VirtualLab多智能体协同系统。来源:https://x.com/james_y_zou/status/1856729107045982607
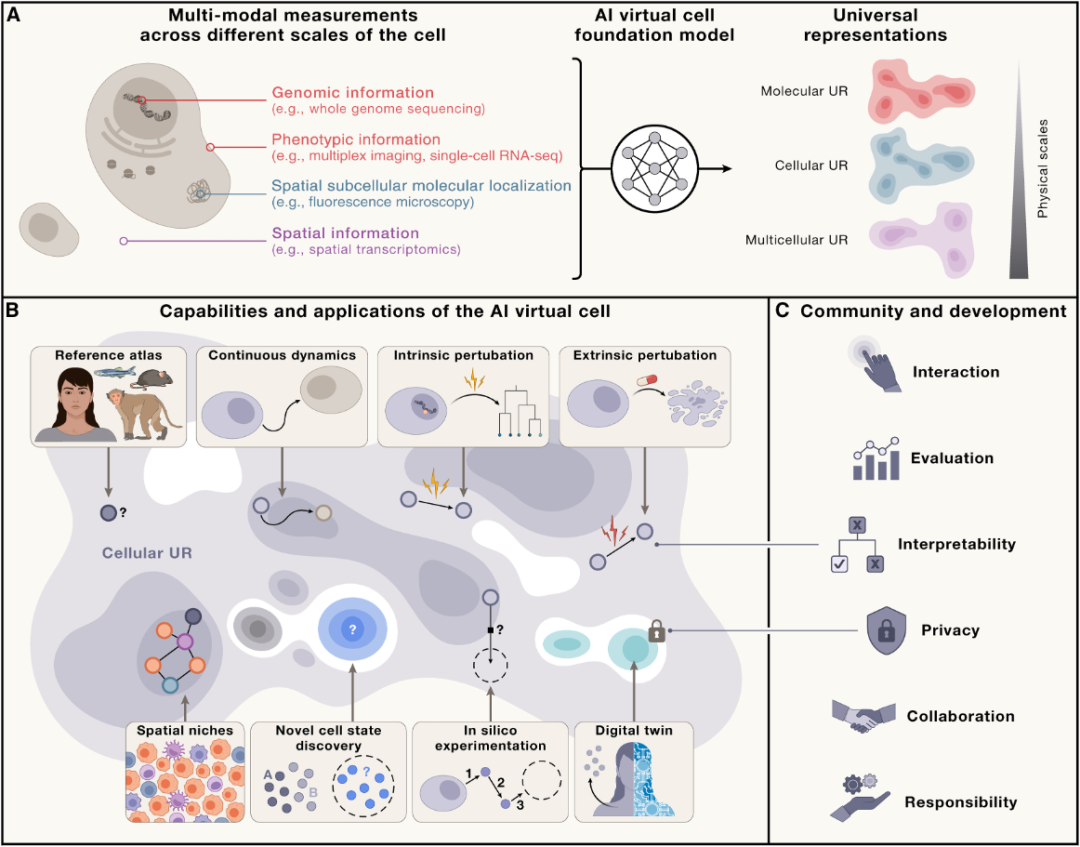
生命大语言模型的蓬勃发展激发了研究者构建人工智能虚拟细胞(AI Virtual Cell,AIVC)[19]的愿景。该虚拟细胞通过使用多种模型来模拟分子、细胞及组织的动态行为,进而解析其运作机制。得益于过去二十年全球科研项目的积累,如人类基因组计划(Human Genome Project)、人类细胞图谱(Human Cell Atlas)、癌症基因组图谱(Cancer Genome Atlas)、DNA元件百科全书(ENCODE)、人类蛋白质图谱(Human Protein Atlas)等,训练机器学习模型已不存在数据瓶颈。虽然构建人工智能虚拟细胞的具体实现时间表尚不明确,但该项目的变革潜力已获40余位顶尖科学家的认可:人工智能虚拟细胞将彻底革新科研范式,有望在生物医学研究、精准医疗、药物研发、细胞工程及可编程生物学等领域带来突破性进展。
全球细胞图谱计划丨来源:How to build the virtual cell with artificial intelligence: Priorities and opportunities
Bunne, Charlotte et al.Cell, Volume 187, Issue 25, 7045 – 7063
随着多领域研究齐头并进,我们正目睹生命科学基础模型的井喷式发展——这些模型正在深化人类对生物分子结构、功能、演化及相互作用的理解,并揭示细胞内部运作的深层机制。这种突破甚至让部分学者将数字生物学等同于工程实践。正如英伟达(NVIDIA)首席执行官黄仁勋所说:“在人类历史首次,生物学有机会成为工程实践,而不仅仅是科学探索”。
谷歌深度学习的Demis Hassabis进一步阐述了这一观点:“我称之为工程科学,因为与自然科学不同,工程科学必须先行人工合成目标构件,而后才能运用科学方法解构该实体并认识其组成部分。”
然而,正如Philip Ball在他的书《生命如何运作:新生物学的用户指南》(How Life Works: A User’s Guide to the New Biology)中指出的,生命的语言远比我们所理解的要复杂得多。他强调,“将生命比作机器、机器人或计算机,是对生命的低估。”他进一步写道,“生命是级联反应过程,每个过程都具有独立的结构完整性与功能自主性,其运作逻辑在非生命领域没有对应范式。”这一论断或许成立,但当前人工智能系统已初步展现解构生命语言极端复杂性的能力——更深刻的突破正在酝酿之中。
1.AlphaFold,能够以原子级精度预测蛋白质结构 Jumper, J., Evans, R., Pritzel, A. et al. Highly accurate protein structure prediction with AlphaFold. Nature 596, 583–589 (2021). https://doi.org/10.1038/s41586-021-03819-2
2.关于基础模型的机遇与风险 https://crfm.stanford.edu/assets/report.pdf
3.一种由DeepMind开发的深度学习模型,能够高精度预测蛋白质的三维结构 Eric Nguyen et al. ,Sequence modeling and design from molecular to genome scale with Evo.Science386,eado9336(2024).DOI:10.1126/science.ado9336
4.AlphaFold 3是一种新型AI模型,能够以前所未有的精度预测所有生物分子的结构及其相互作用 Abramson, J., Adler, J., Dunger, J. et al. Accurate structure prediction of biomolecular interactions with AlphaFold 3. Nature 630, 493–500 (2024). https://doi.org/10.1038/s41586-024-07487-w
5.Boltz-1开源深度学习模型,能够以与AlphaFold3相当的精度预测生物分子复合物的3D结构 Boltz-1 Democratizing Biomolecular Interaction Modeling
Jeremy Wohlwend, Gabriele Corso, Saro Passaro, Mateo Reveiz, Ken Leidal, Wojtek Swiderski, Tally Portnoi, Itamar Chinn, Jacob Silterra, Tommi Jaakkola, Regina Barzilay
bioRxiv 2024.11.19.624167; doi: https://doi.org/10.1101/2024.11.19.624167
6.MassiveFold,一种优化和可定制的AlphaFold版本 Raouraoua, N., Mirabello, C., Véry, T. et al. MassiveFold: unveiling AlphaFold’s hidden potential with optimized and parallelized massive sampling. Nat Comput Sci 4, 824–828 (2024). https://doi.org/10.1038/s43588-024-00714-4
7.EVOLVEpro的计算方法 Kaiyi Jiang et al. ,Rapid in silico directed evolution by a protein language model with EVOLVEpro.Science387,eadr6006(2025).DOI:10.1126/science.adr6006
8.PocketGen深度生成模型,能够高效生成蛋白质结合口袋的残基序列和原子结构 Zhang, Z., Shen, W.X., Liu, Q. et al. Efficient generation of protein pockets with PocketGen. Nat Mach Intell 6, 1382–1395 (2024). https://doi.org/10.1038/s42256-024-00920-9
9.PIONEER深度学习框架 Xiong, D., Qiu, Y., Zhao, J. et al. A structurally informed human protein–protein interactome reveals proteome-wide perturbations caused by disease mutations. Nat Biotechnol (2024). https://doi.org/10.1038/s41587-024-02428-4
10.AbMAP的迁移学习框架 R. Singh, C. Im, Y. Qiu, B. Mackness, A. Gupta, T. Joren, S. Sledzieski, L. Erlach, M. Wendt, Y. Fomekong Nanfack, B. Bryson, & B. Berger, Learning the language of antibody hypervariability, Proc. Natl. Acad. Sci. U.S.A. 122 (1) e2418918121, https://doi.org/10.1073/pnas.2418918121 (2025).
11.RNA结构预测工具 RhoFold+ Shen, T., Hu, Z., Sun, S. et al. Accurate RNA 3D structure prediction using a language model-based deep learning approach. Nat Methods 21, 2287–2298 (2024). https://doi.org/10.1038/s41592-024-02487-0
12.RhoDesign深度学习平台 Wong, F., He, D., Krishnan, A. et al. Deep generative design of RNA aptamers using structural predictions. Nat Comput Sci 4, 829–839 (2024). https://doi.org/10.1038/s43588-024-00720-6
13.GET(General Expression Transformer)通过分析213个人类胎儿和成人细胞类型的染色质可及性和DNA序列,揭示基因调控的语法,从而准确预测基因活性。Fu, X., Mo, S., Buendia, A. et al. A foundation model of transcription across human cell types. Nature 637, 965–973 (2025). https://doi.org/10.1038/s41586-024-08391-z
14.基于多物种基因组比对的DNA语言模型,预测全基因组变异的影响
Benegas, G., Albors, C., Aw, A.J. et al. A DNA language model based on multispecies alignment predicts the effects of genome-wide variants. Nat Biotechnol (2025). https://doi.org/10.1038/s41587-024-02511-w
15.MethylGPT,预测DNA甲基化值并应用于年龄预测和疾病诊断
MethylGPT: a foundation model for the DNA methylome
Kejun Ying, Jinyeop Song, Haotian Cui, Yikun Zhang, Siyuan Li, Xingyu Chen, Hanna Liu, Alec Eames, Daniel L McCartney, Riccardo E. Marioni, Jesse R. Poganik, Mahdi Moqri, Bo Wang, Vadim N. Gladyshev
bioRxiv 2024.10.30.621013; doi: https://doi.org/10.1101/2024.10.30.621013
16.CpGPT,从有限的输入数据重建全基因组甲基化图谱,并在年龄预测和死亡风险评估等任务上表现出色
CpGPT: a Foundation Model for DNA Methylation
Lucas Paulo de Lima Camillo, Raghav Sehgal, Jenel Armstrong, Albert T. Higgins-Chen, Steve Horvath, Bo Wang bioRxiv 2024.10.24.619766; doi: https://doi.org/10.1101/2024.10.24.619766
17. SyntheMol 用生成模型设计抗生素结构
Swanson, K., Liu, G., Catacutan, D.B. et al. Generative AI for designing and validating easily synthesizable and structurally novel antibiotics. Nat Mach Intell 6, 338–353 (2024). https://doi.org/10.1038/s42256-024-00809-7
18. SCimilarity细胞图谱基础模型,能够高效地搜索和比较人类体内相似的单细胞状态
Heimberg, G., Kuo, T., DePianto, D.J. et al. A cell atlas foundation model for scalable search of similar human cells. Nature (2024). https://doi.org/10.1038/s41586-024-08411-y
How to build the virtual cell with artificial intelligence: Priorities and opportunities Bunne, Charlotte et al. Cell, Volume 187, Issue 25, 7045 – 7063
大模型与生物医学:
AI + Science第二季读书会
生物医学是一个复杂且富有挑战性的领域,涉及到大量的数据处理、模式识别、理论模型建构和实验验证等问题。AI基础模型的引入,使得我们能够从前所未有的角度去观察和理解这个领域的问题,加速科学研究的步伐,提高医疗服务的效率和效果。这种交叉领域的合作,标志着我们正在向科技与生物医学深度融合的新时代迈进,对于推动科学研究、优化医疗服务、促进人类健康有着深远的影响。
集智俱乐部联合西湖大学助理教授吴泰霖、斯坦福大学计算机科学系博士后研究员王瀚宸、博士研究生黄柯鑫、黄倩,华盛顿大学博士研究生屠鑫明,共同发起以“大模型与生物医学”为主题的读书会,共学共研相关文献,探讨基础模型在生物医学等科学领域的应用、影响和展望。读书会已完结,现在报名可加入社群并解锁回放视频权限。
详情请见:
大模型与生物医学:AI + Science第二季读书会启动
6. 探索者计划 | 集智俱乐部2025内容团队招募(全职&兼职)